转载自:生信草堂|原创作者:土豆力
该篇文章为数据库系列文章的第二篇,文章由哈尔滨医科大学Chunquan Li’s Lab 8月发表在Briefings in Bioinformatics杂志上文章题目为TRCirc: a resource for transcriptional regulation information of circRNAs,主要关注于circRNA的调控信息检索及可视化,下载地址http:// doi.org/10.1093/bib/bby083;
在线网址为http://www.licpathway.net/TRCirc。
研究背景
circRNA由于其独特的拓扑结构及组织时空特异性表达受到广泛关注,自ciRS-7研究中报道了circRNA具有海绵(sponges)功能后,越来越多的circRNA的调控模式逐步揭开。最新的研究显示,转录因子Twist1能够选择性的调控circRNA Cul2而不是其来源基因,显示转录因子能够调控多个类型的基因,但是其中的调控机制仍不清楚。借助于高通量测序技术的普及,转录组测序(RNA-seq)结合染色质免疫沉淀测序(ChIp-seq)联合分析有助于揭开转录因子在调节非编码RNA的过程的机制。因此,作者开发了TRCirc在线工具,允许研究人员轻松搜索和浏览TF-circRNA调控关系和其他相关信息,包括表达,H3K27ac信号和甲基化水平。
数据库概况
TRCirc的数据内容来源于uniform transcription factor binding site(TFBS) data sets, ChIP-seq data sets involving H3K27ac(ENCODE), RNA-seq data sets and 450 K arrays.通过流程分析,目前发布的TRCirc包含超过100种细胞类型,涉及690个TFBS,36个H3K27ac,54个RNA-seq和61个450k数据集,包含92375个来源于circRNA和161个转录因子,涵盖151个细胞系中circRNA相关的遗传和表观遗传信息,包括DNA甲基化和超增强子H3K27ac信号和表达情况。细胞类型包括永生化细胞系,原代细胞,干细胞,体外分化细胞和诱导多能干细胞系。
用户向导
1. 搜索特定circRNA获得所有已报道的调控信息
Advanced Search 支持用户搜索并浏览单个circRNA调控相关的转录因子及对应的甲基化探针ID。通过选择(i)物种和细胞类型和(ii)circRNA的细胞系和调节区(例如上游30000bp和起始位点下游2000bp)并输入感兴趣的特定circRNA(图3A),获得TF-circRNA调节关系,H3K27ac信号,平均甲基化水平等信息(图3G)。
2. 搜索和浏览转录因子或细胞系过滤获得TF-circRNA调控关系
通过选择(i)circRNA的种类和调控区域(例如上游20 000 bp和起始位点下游1000 bp)和(ii)TF或细胞系(图3B和C),获得该条件下搜索其调节区域中TF峰(peak)对应的circRNA(图3H和I)。
3. 同时支持选择(i)circRNA的种类和调控区域(例如,其起始位点上游20 000 bp和下游1000 bp)和(ii)感兴趣的细胞系和输入cRNA(图3D),获得特定细胞系中的circRNA的甲基化状态(图3J)、超级增强子和增强子等信息(图3E和K)。内置的JBrowse浏览器实现相关的转录因子结合位点和超级增强子可视化,同时包含单核苷酸多态性(SNP)等信息。并提供可视化工具在线构建TF–circRNA互作网络(Cytoscape web tool),并支持获得相关circRNA的表达数据(RPKM)统计图。
4. TRCirc允许下载数据包括:TF-circRNA调控关系对,表达水平,甲基化水平,H3K27ac信号和与特定circRNA相关的超级增强子信息(图3M)。
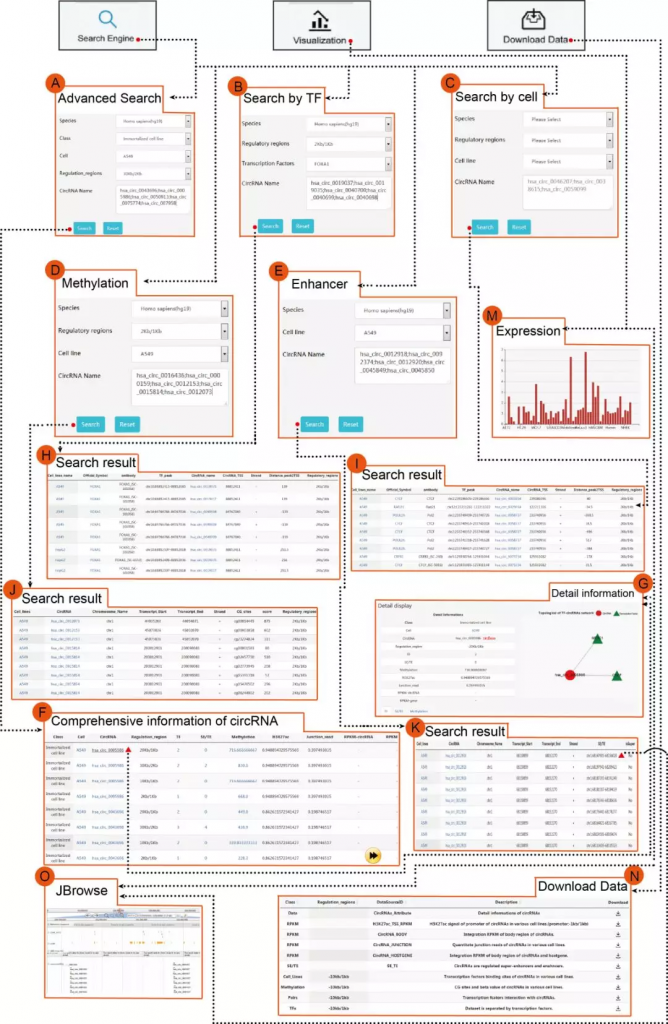
Figure 3. A comprehensive view of TRCirc search and query results. Panels (A), (B), (C), (D) and (E) are search interface of comprehensive, TF, cell line, methylation and enhancer, respectively. Common options include ‘Species’, ‘Regulatory regions’ and ‘CircRNA Name’; ‘Class’ and ‘Cell’ as two options for search interface of comprehensive; ‘Transcription Factors’ as one option for search interface of TF; and ‘cell line’ as one option for search interface of cell line and methylation. In particular, ‘Regulatory regions’ not as one option for search interface of enhancer. Panel (F) is the result table of comprehensive search. ‘TF’ represents the number of TFs that regulate a specific circRNA.‘SE/TE’ represents the number of enhancers associated with specific circRNA.‘Methylation’ represents the mean methylation level of specific circRNA. (G) Details of a selected circRNA from the results table. Panels (H), (I), (J) and (K) are result table of TF, cell lines, methylation and enhancer search, respectively. Panel (L) is the illustration of data visualization.(M) Download page. (N) JBrowse.
小 结
TRCirc整合了ENCODE数据库上的ChIP-seq, RNA-seq 和 450 K array data数据并联合circBase上人类相关的circRNA信息用于分析circRNA转录调控。同时提供搜索,浏览及可视化工具,快速搜索及过滤特定circRNA详细调控信息。包括表达水平,甲基化水平,调节区中的H3K27ac信号及超级增强子,并支持下载。目前只支持人类数据搜索,为circRNA下游分析提供一个有效筛选工具。





来第一个抢占沙发评论吧!