转自:生信菜鸟团(微信公众号)
上期回顾
上一期较为完整地介绍了 find_circ 的原理以及实战,主要使用 bowtie2 将转录组数据比对到参考基因组上,然后使用 python脚本获取未比对上序列两端anchors,再次比对后,补齐序列后判断方向及剪切位点,最后根据关键词筛选及合并获得候选序列。
但是,纷繁的生物信息学,给出了很多种不同的解法,我们应该怎么去判断或者快速展开数据分析呢。别担心,有一些前辈帮我们先走出了一大步,我们来看看他在circRNA程序鉴定上给出的最优解是什么。
分析材料
- 数据来源
数据下载于SRA (SRR444655 vs SRR444975, SRR444974 vs SRR445016),并添加 AGATCGGAAGAGC 序列,hg19基因组作为参考基因组。 - 程序选择及前提
总共对比11种识别程序 (表1),使用默认参数。每一个程序鉴定的circRNA必须至少包含3个reads;KNIFE 检验值在0.9以下将被舍弃,CIRCexplorer中内含子circRNA舍弃;舍弃所有来源于线粒体基因组的circRNA;CIRCexplorer2, KNIFE and MapSplice 三者比较无参识别。
结果讨论
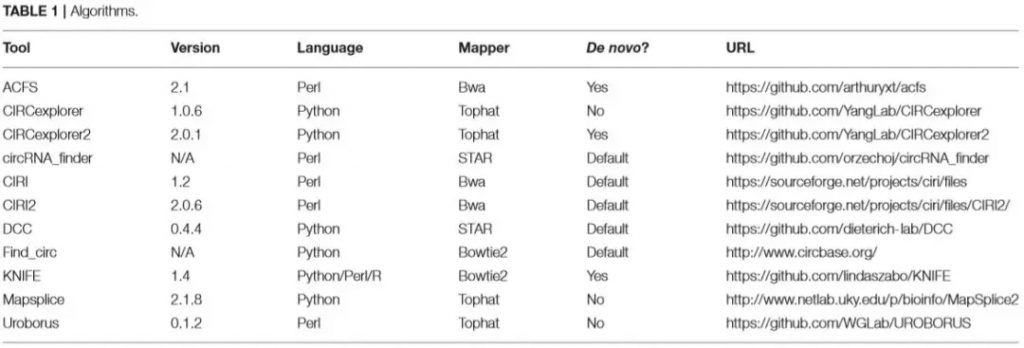
- RNAse R Resistance
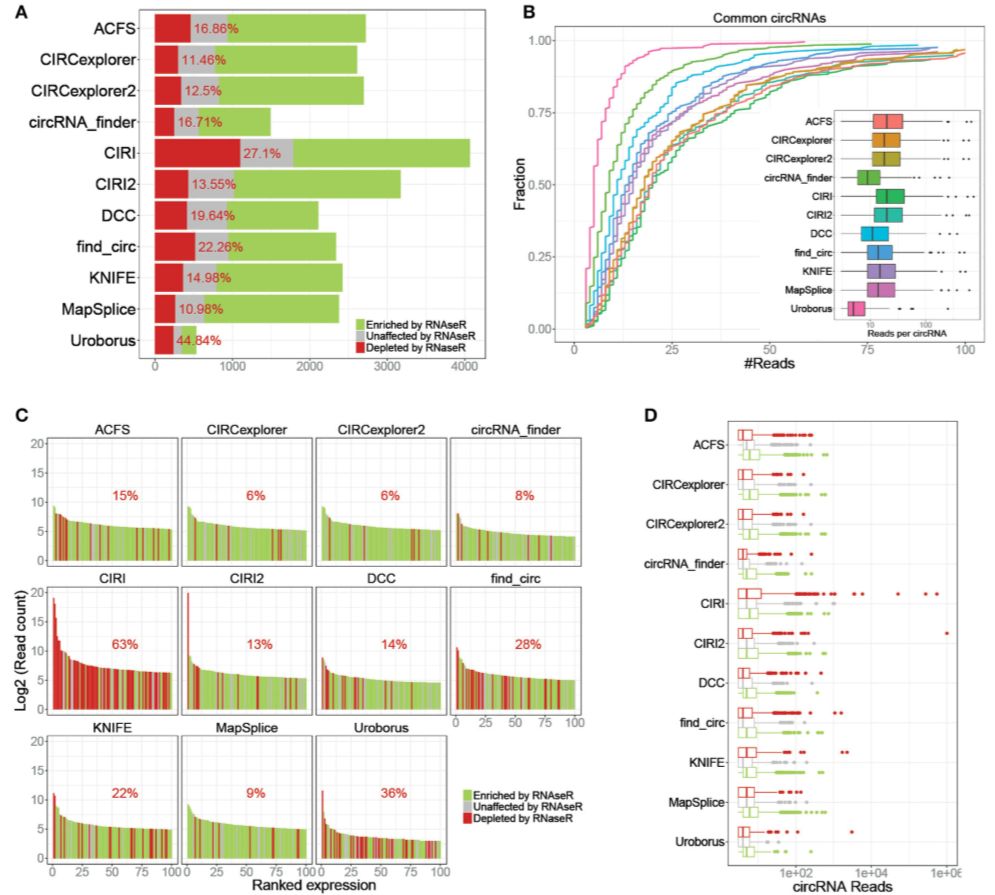
首先作者对不同方式处理文库获得的转录组数据进行比较。结果显示 RNAse R 处理后的数据能够大量减少鉴定的假阳性,且对TOP100的circRNA的影响最大 (Fig 1)。
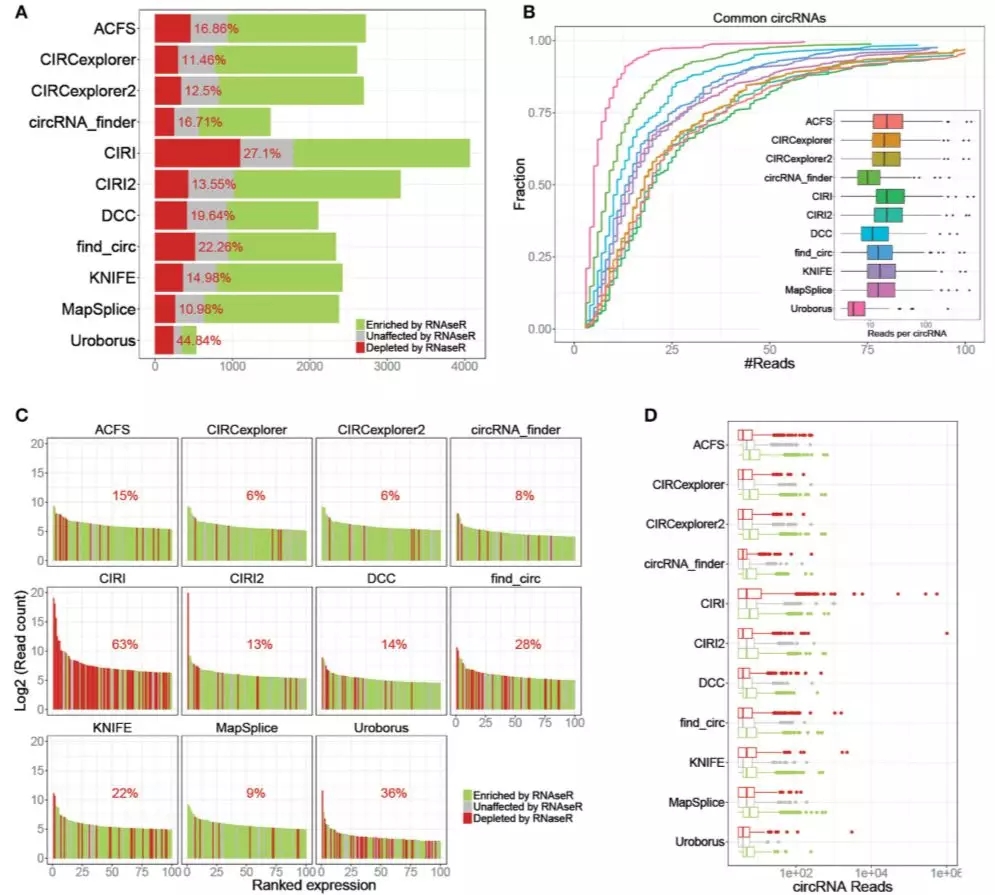
- Raw vs. Trimmed Reads
不同的程序鉴定反向剪切读段的方法不尽相同,因此read的质量以及3‘ 接头序列对这些程序来说至关重要。作者对比了raw data及clean data的鉴定结果,处理后的数据将提升大部分的程序都获得了更多的circRNA预测数量 (0–27%),适度提高了敏感度,但同时也增加了预测的假阳性 (13–67%, 12–48% using raw sequence reads)。能够确定的是,KNIFE, circRNA_finder and Uroborus,使用处理后将更有利于识别 (Fig 2)。这里需要注意的一点是:这些评估结果使用的数据为高质量的测序结果,因此在对待低质量的数据时需要更多的考虑对raw data进行过滤。
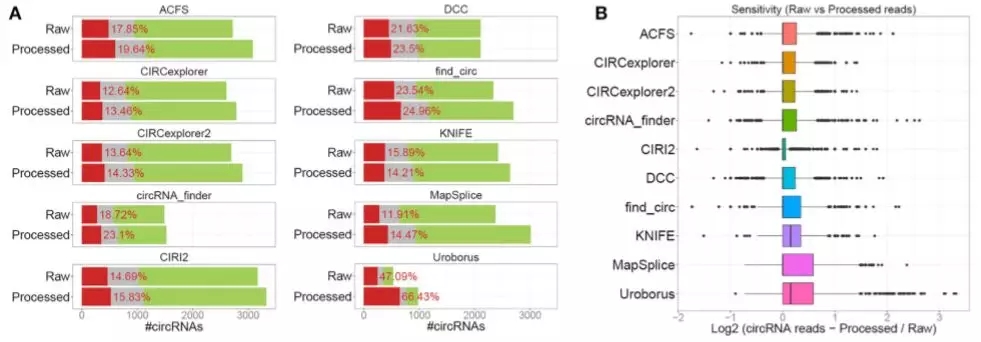
- De Novo Prediction
作者使用RNAseR 处理的数据对三个 (ACSF, CIRCexplorer2 and KNIFE) 支持无参预测程序进一步比较,使用 De Novo Prediction时,三者表现更低效,ACSF, CIRCexplorer2虽然并没有很明显差距,但是 KNIFE 不依赖注释时的结果天差地别,因此 KNIFE 不适合无参的方法。而CIRI2 默认的无参算法表现的更佳。有参分析过程会避免一些不真正来源于有注释剪切位点的 bona fide circRNA,但是对于一些并没有良好注释的物种来说,该结论并不适用 。 - Combining Prediction Algorithms
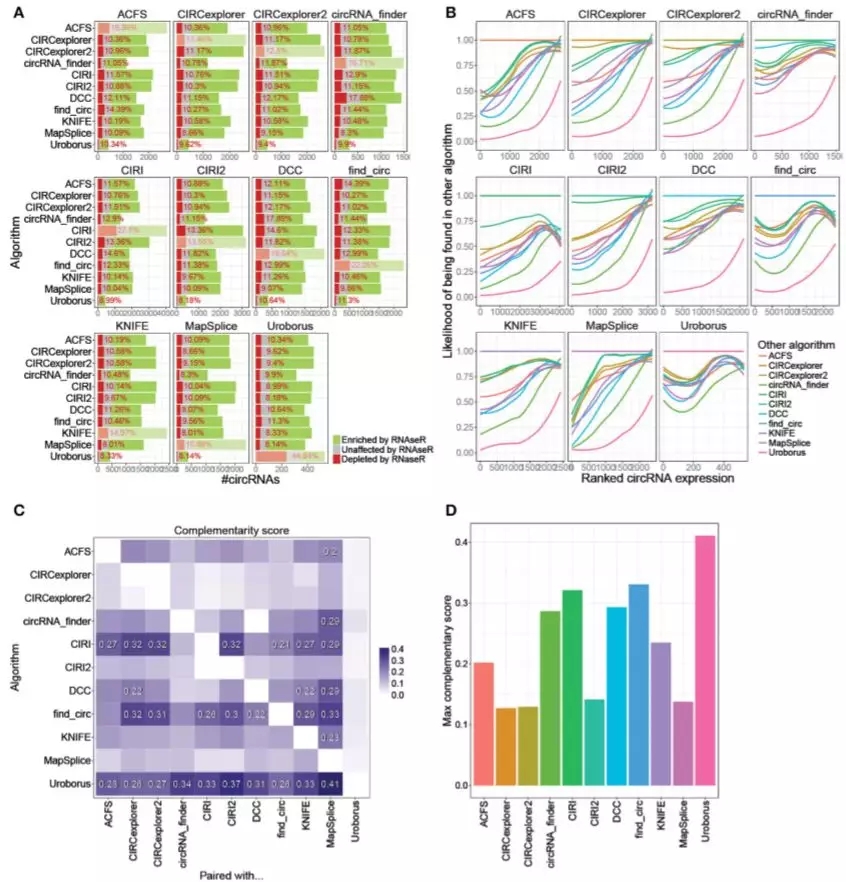
绝大多数的算法都支持高表达circRNA的真阳性,但同时也会过滤掉一些低相关度预测的结果。在结合多种算法的过程中,需要注意辨别不同算法间的差异,选择不同预测模型的算法将降低假阳性的概率。同时作者提供一个计算方法可以粗略估计两种算法的互补程度,有兴趣的同学可以详看过程,引用如下:
To establish general guidelines for combination of algorithms, a rough measurement of complementarity was established:
- First, for each algorithm, an index of true positives (iTP), reflecting the fraction of preserved circRNAs with RNaseR resistance after conjoining with any other algorithm, was determined
- Then, similarly, the fraction of discarded true negatives was calculated, which is the inverse fraction of shared true negatives.
- Finally, to evaluate the overall conjoining effect of any two algorithms, a Complementary score is proposed (Complementary score = (iTPxiTN)∧2) that scores the achieved benefit of pairing one algorithm with any other algorithm.
我们来看结果,CIRI, find_circ, and Uroborus更适合于与其他程序结合使用,而MapSplice 看似是一个首选的补充程序。要注意的是,CIRI2虽然整体表现很好,但是仅依靠一种程序获得高丰度的候选者往往会带来假阳性 (Fig.4)。
- Reproducibility
作者使用已报道文献中的数据使用不同的鉴定程序进行可重复性实验,对比发现, MapSplice, the CIRCexplorers and CIRI2表现最好,CIRI2在无参预测中非常出色。而多个程序的组合使用将会得到更准去的预测。



来第一个抢占沙发评论吧!