时下最火的RNA是谁?那当然非ncRNA莫属了! ncRNA带着它的freestyle强势来临了!借着这freestyle小编今天就来和您聊聊circRNA那些你不得不知道的事,快跟上我的脚步吧!
Q:我们为什么要关注circular RNA?
A:环状RNA(circRNA)是一类具有闭合环状结构的非编码RNA,其闭环结构可以逃逸RNA酶的降解,因此比线性RNA更稳定。已有的研究表明,circRNA在脑发育神经细胞分化、精神疾病(如阿尔茨海默症)、肿瘤发生与发展中均发挥重要作用。circRNA的研究为非编码RNA的研究开拓了一个新的领域。
Q:如何做环状RNA的序列及功能验证?
A:今天为大家推荐几种circRNA的功能验证策略,主要包括:circRNA定量验证(qPCR法)、Northern blot验证、过表达(正向验证)、基因沉默(反向验证)。
1circRNA定量验证(qPCR法)
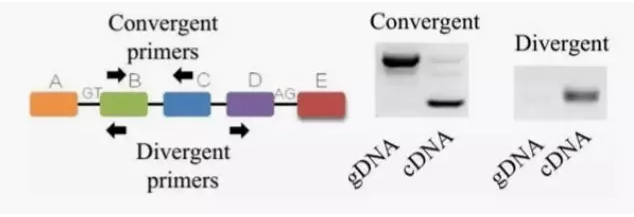
常规的线性mRNA在qRT-PCR验证是采用前后设计探针的方式,而circular RNA验证时则采用和线性mRNA分子引物方向相反的方式(Divergent PCR),以证明circRNA确实是存在和差异表达。
qPCR线性RNA和环状RNA设计原理及扩增产物图
circRNA引物设计相比线性RNA比较麻烦。因此,很多人苦于对调取circRNA全长及验证circRNA的成环性。今天向大家分享circRNA调取全长序列及引物的设计方法,希望能帮助各位顺利开展circRNA的研究。
circRNA全长调取及引物设计方法
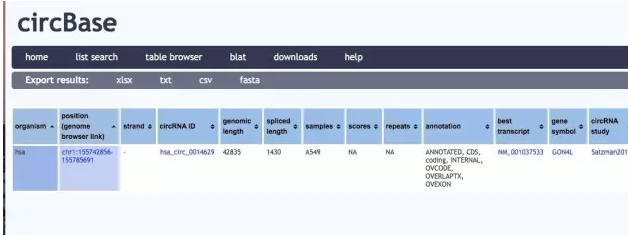
1)在研究circ RNA时,我们主要利用cirbase数据库。
通过cirbase数据库(http://www.circbase.org/)获得circRNA的序列,例如hsa_circ_0014629:
2)将网页中的 circ sequence 选项更改为“spliced”,再点击 “search”,即可下载circRNA的全长序列信息:
3)点击fasta下载序列,可以得到下图的序列:
4)针对上述序列,即可设计circRNA的全长序列调取引物,设计方法与常规基因引物设计完全一致。取首尾各150bp进行拼接,末尾150bp作为拼接后的首序列,如下:

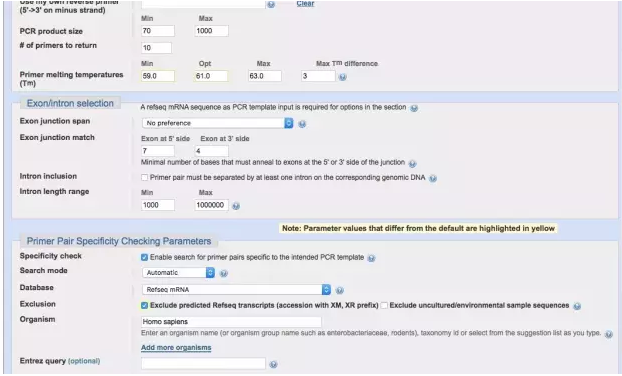
5)将该部分300bp用primer-blast进行引物设计,具体参数如下:
6)选择引物对。注:PCR产物包含back-splicing位点

2Northern blot验证circRNA序列
在Northern blot方法验证RNA序列时需要注意的是探针的设计是事关杂交实验成败的关键因素,对于exon环化circRNA,设计的探针要求跨越back splice junction位点;对于含内含子环化circRNA,除了跨back splice junction位点设计探针外,还可在内含子区域设计探针。
验证步骤:
A. 对total RNA去DNA、去rRNA、去线性RNA(RNAseR/H消化)处理,与探针杂交;
B. Total RNA与探针杂交;
C. 基因组DNA与探针杂交。
以上A、B、C同时进行,共同验证。
3过表达——正向验证
circRNA过表达的原理主要是基于circRNA的成环机理。形成circRNA的外显子侧翼序列含有互补的Alu元件。基于circRNA侧翼Alu序列特征,PCR扩增含侧翼Alu序列的目标DNA序列(注意扩增的模板就是DNA),随后依据对应限制性内切酶位点进行酶切,进而连接载体,转染进对应细胞样本。设计引物扩增产物后进行片段回收,通过sanger测序验证转染效率,或构建共表达载体(带GFP标签)通过荧光判断转染效率,最后利用divergent primer验证circRNA过表达情况。
过表达建议:扩增包含circRNA侧翼Alu序列或内部碱基互补序列的目标区域。有研究表明侧翼上下游1kb处过表达效果更明显。
4基因沉默——反向验证
通过siRNA干扰封闭circRNA的功能。针对外显子环化circRNA,可在back splice junction位点前后设计siRNA;针对内含子环化circRNA,除了back splice junction位点前后序列设计siRNA以外,也可针对内含子序列设计siRNA;每个siRNA设计对应对照,对照siRNA设计要求:back splice junction位点一端互补配对,另一端错配(阴性对照)。
siRNA沉默circRNA功能
通过小编今天的介绍,大家是不是对circRNA定量引物的设计以及功能验证有了更深的了解呢,希望可以为各位科研大大的研究助上一臂之力!
参考文献
[1] Li Z, Huang C, Bao C, et al. Exon-intron circular RNAs regulate transcription in the nucleus[J]. Nature Structural & Molecular Biology, 2015, 22(3):256.
[2] Rybak-Wolf A, Stottmeister C, Glažar P, et al. Circular RNAs in the Mammalian Brain Are Highly Abundant, Conserved, and Dynamically Expressed[J]. Molecular Cell, 2015, 58(5):870-85.
[3] Conn, Simon , Pillman, et al. The RNA Binding Protein Quaking Regulates Formation of circRNAs.[J]. Cell, 2015, 160(6):1125-34.
[4] Jeck W R, Sharpless N E. Detecting and characterizing circular RNAs.[J]. Nature Biotechnology, 2014, 32(5):453-61.
来源:诺禾致源






来第一个抢占沙发评论吧!