在芯片测序类文章能发几分一文中,我们分析了17年的芯片测序类文章的整体情况,结果表明circRNA的文章还是蛮坚挺的。
目前circRNA可用的数据库同它的相关文章一样还比较少,今天给大家扒一扒circRNA有哪些好用的数据库。
1、circbase
在circBase(http://www.circbase.org/cgi-bin/listsearch.cgi)中,我们可以通过circRNA名称(hsa_circ_0010153),转录本名称(NM_133494),染色体定位(chr1:12359258-12382803)检索circRNA的信息(序列,定位,gene_symbol等信息),你也可以直接检索mRNA相关(TP53)或是生物学过程相关(apoptosis)的circRNA。
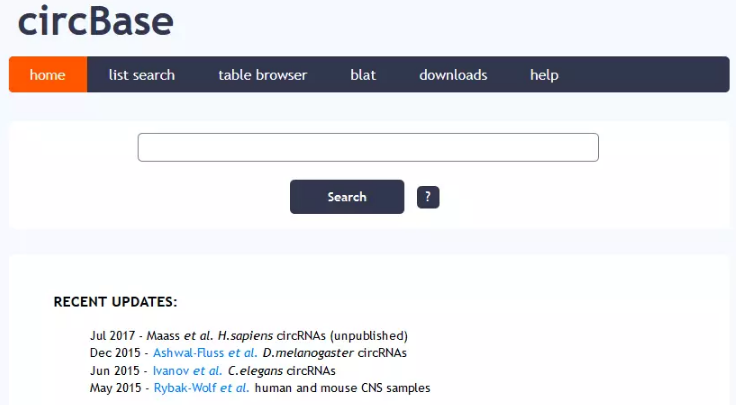
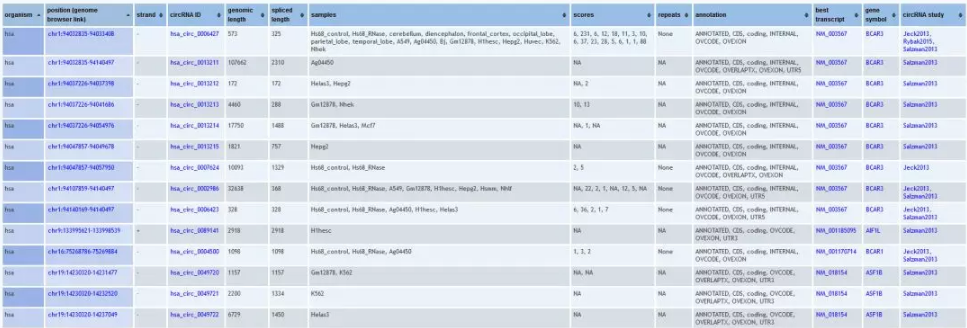
circBase有blat功能可以进行序列比对,用于了解基因的物种保守性,不过物种较少
推荐使用UCSC(http://genome.ucsc.edu/)的blat功能
其实circBase的序列信息还是源于UCSC
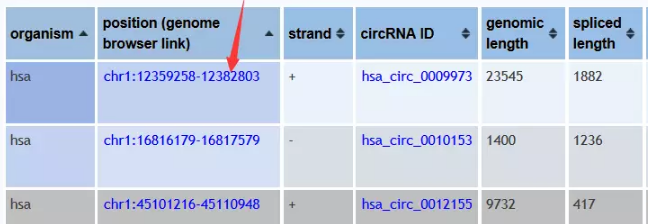
但是在UCSC中直接用hsa_circ_0010153直接搜,还偏偏搜不出下面这样的结果
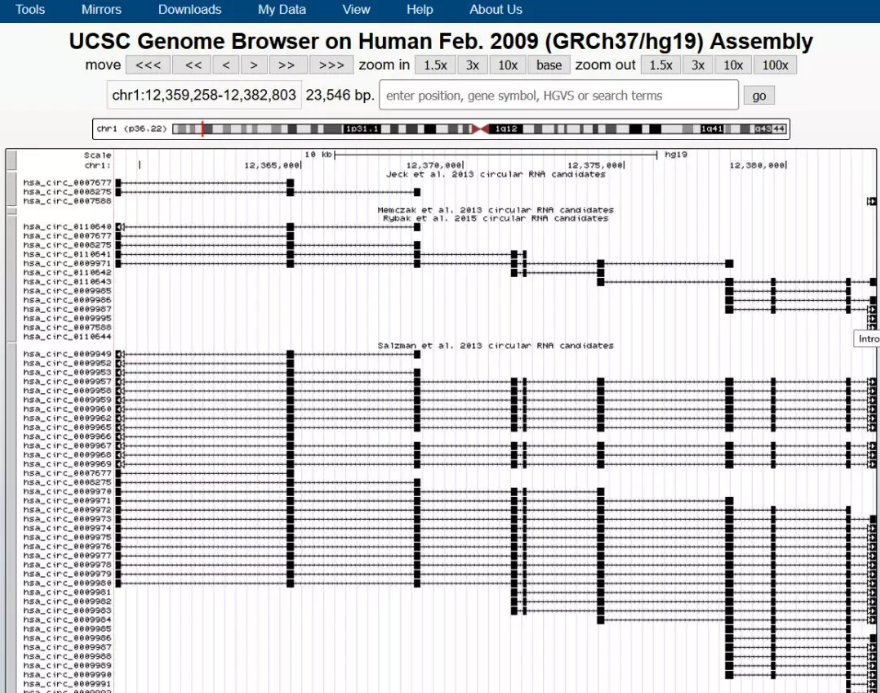
点开一条序列,即可获取该条序列的信息
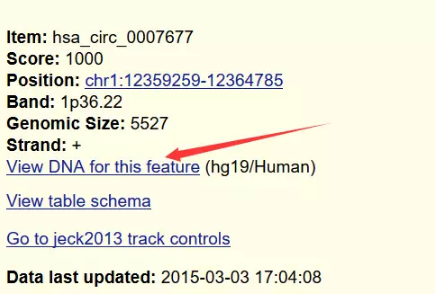
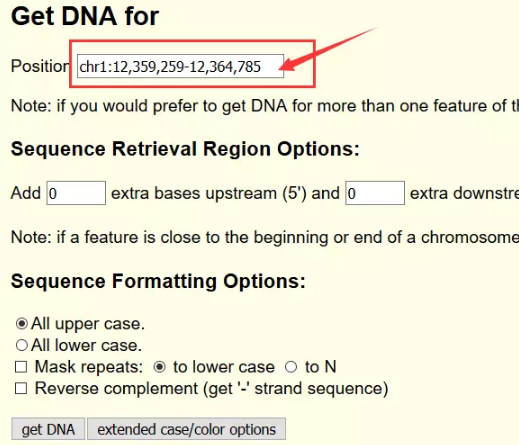
前面在circBase中,我们已经获取了circRNA的位置信息了,所以就不需要再一个个点开查看基因序列了,直接在UCSC中输入位置信息即可获得完整的基因序列。
2、RegRNA2.0
那我们获取了基因序列可以干嘛呢?第一反应当然是预测miRNA咯~
在RegRNA 2.0(http://regrna2.mbc.nctu.edu.tw/)中,我们可以用基因序列做很多事情,当然也包括预测miRNA。
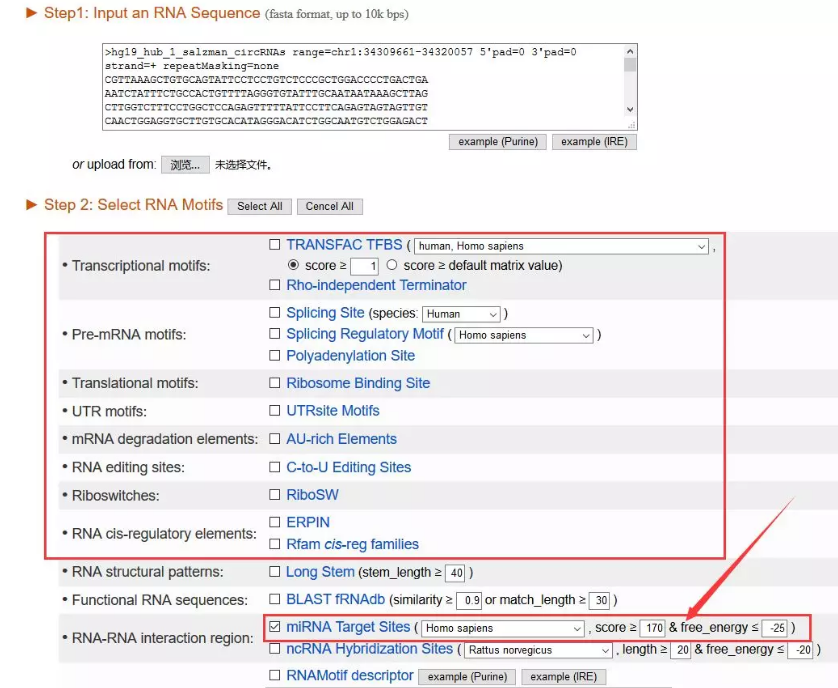
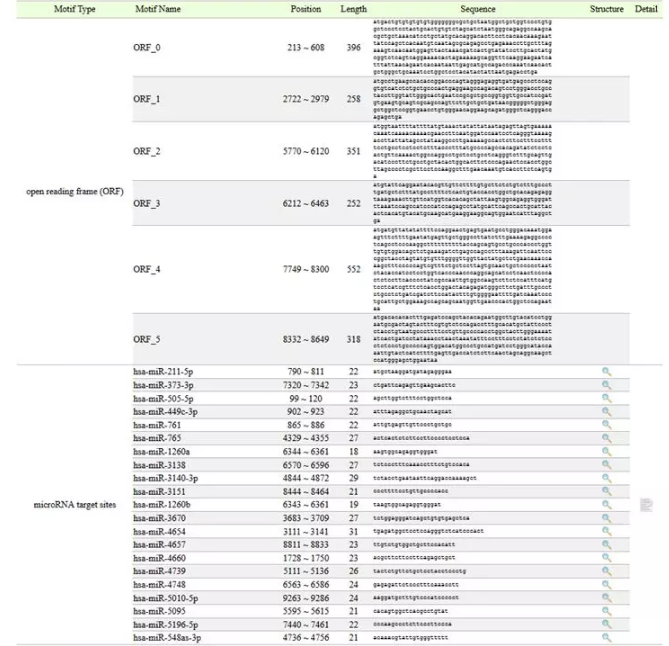
miRNA预测结果如下

3、circlnteractome
嫌上面这个预测circRNA的miRNA太麻烦?那你可以试试CircInteractome(https://circinteractome.nia.nih.gov/)
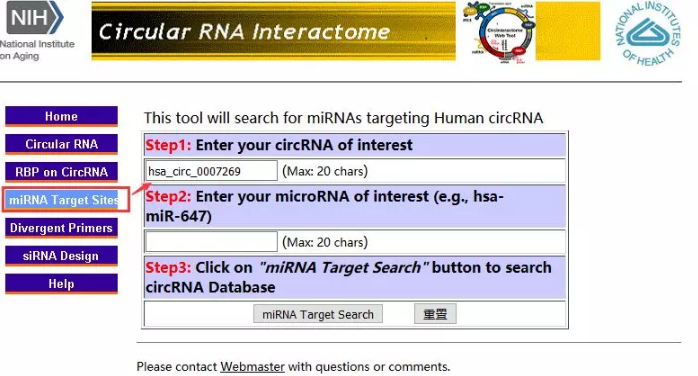
就是这么简单粗暴
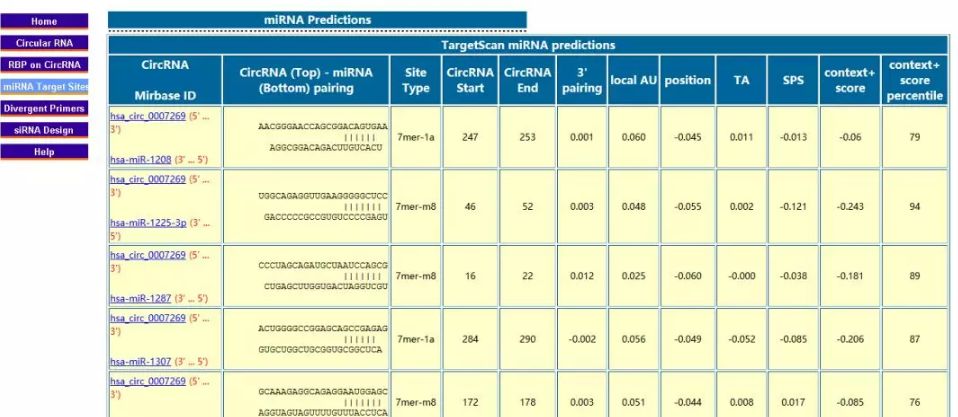
以上的检索结果如果都不能让你满意,你可以试试曲线救国的方式,用circRNA的gene sybmol来搜索相关的miRNA,比如hsa_circ_0009973的gens symbol是
VPS13D。不过这样一来你得确认circRNA与miRNA结合区域在mRNA和circRNA序列中都是存在的,如果结合区域由于剪切方式不同在circRNA中被剪没了那就玩不起来了。此外由于circRNA是环状的,所以在和miRNA结合时,自由能等等参数肯定会由于分子构象发生改变,所以预测的准确性也会打折扣。
文章:小张聊科研





很多都上不去了