文章解读: |
Expression profile of circular RNAs in infantile hemangioma detected by RNA-Seq
RNAseq研究婴儿血管瘤circRNAs表达谱
期刊:MEDICINE 影响因子:1.804
发表单位:南京市妇幼保健院医疗美容科
1 摘要
背景:circular RNAs(circRNAs)是一类新出现的广泛存在的非编码RNAs,在各种生物学过程中发挥着重要作用。然而,对婴儿血管瘤infantile hemangioma(IH)中circRNAs的特征和功能并不了解。
方法:本研究,我们用RNAseq技术和circRNAs预测研究circRNAs在婴儿血管瘤组织和正常配对皮肤组织中circRNAs的特征。对个别circRNAs进行RT-PCR验证。
结果与结论:婴儿血管瘤相比正常配对皮肤组织中,共发现9811个circRNAs,249个差异表达,包括124个上调,125个下调。qPCR验证了一些差异表达circRNAs(hsa_circRNA001885 and hsa_circRNA006612)。对差异表达circRNAs进行GO和Pathway分析,发现涉及许多生物学过程如binding, protein binding, gap junction, and focal adhesion。使用miRanda预测了几个circRNAs的microRNAs。本研究我们发现circRNAs在婴儿血管瘤中潜在的重要性,及对于治疗的潜在价值。
缩略语:ceRNAs = competitive endogenous RNAs, circRNAs = circular RNAs, GAPDH = glyceraldehyde 3-phosphate dehydrogenase, GO = gene ontology, IH = infantile hemangioma, KEGG = Kyoto Encyclopedia of Genes and Genomes, lncRNAs = long noncoding RNAs, miRNA = microRNA, MREs = microRNA response elements, mRNAs = messenger RNAs, qRT-PCR = realtime quantitative PCR, RNA-Seq = RNA sequencing, SD = standard deviation.
2 材料方法
2.1 伦理声明
本研究获得南京医科大学附属妇产科医院医学伦理委员会批准。婴儿血管瘤患者在我院接受外科治疗,手术获取其血管瘤组织和正常配对皮肤组织,已获得父母知情同意。
2.2 组织样品
三个不同病人获取增殖毛细血管血管瘤和配对正常皮肤组织。常规病理检查判断为增值性血管瘤。收集的皮肤组织立即液氮冻存,以备total RNA提取。病人信息如Table 1。

2.3 total RNA提取
Qiagen miRNeasy Mini Kit (Qiagen, Valencia, CA)提取组织样品total RNA。NanoDrop 2000 (Thermo Fisher, MA)检测RNA纯度,Qubit 3.0 Fluorometer (Life Technologies, CA)对RNA密度进行定量。Agilent 2100 Bioanalyzer (Agilent Technologies, CA)评估RNA完整性。
2.4 文库准备,RNA测序
VAHTS Total RNA-Seq (H/M/R) Library Prep Kit构建转录组测序文库,构建流程如下:首先用特异性探针,RNase H and DNA polymerase I去除rRNA。而后纯化,二价阳离子适宜温度将RNA片段化。反转录酶和自由引物将RNA反转录成第一链cDNA, 而后DNA polymerase I, RNase H, and dNTPs (dUTP, dATP, dGTP, and dCTP)合成第二链。对cDNA添加A,并连接接头序列。选择适宜长度的cDNA用于测序,VAHTS DNA Clean Beads选择文库长度,UDG enzyme消化第二条链,进行PCR扩增,文库纯化。Illumina Hiseq X10 platform测序,模式为PE150。
2.5 质控
自写perl脚本处理raw reads。去除含有接头,“N”超过5%,低质量reads(低质量碱基占整条reads 50%,碱基质量值低于10认为是低质量的碱基)。同时计算clean reads的Q20, Q30, and GC含量。后续分析基于clean reads。
2.7 raw reads过滤
通过去除含有接头,ploy-N 和低质量reads用于后续分析。过滤步骤如下:去除含有接头的reads;去除含有poly-N reads,比例大于5%;去除低质量reads(碱基质量值Q≤10比例大于50%)。后续分析基于clean data。
2.6 比对到参考基因组
参考基因组及注释文件下载于UCSC(hg38)。Bowtie (v2.1.0)构建参考基因组索引,TopHat (v2.1.1)将成对clean reads比对到参考基因组。
2.8 circRNAs预测
使用circRNA_Finder预测circRNAs(https://github.com/bioxfu/circRNAFinder)。首先clean reads使用Bowtie2 (http://bowtie-bio.sourceforge.net/bowtie2/manual.shtml)比对到参考基因组。然后对于未必对的reads,circRNA_Finder鉴定back-splice reads并确认是否是circRNAs。最后,使用circRNAAnno对circRNAs进行注释和统计,获得circRNAs序列用于后续分析。
2.9 circRNAs差异表达分析
circRNAs的表达量用“Transcripts Per Million” (TPM)校正。DESeq基于负二项分布检验进行差异表达分析。差异表达阈值设置为FDR≤0.05 and |log2Fold change| ≥1。
2.10 qPCR验证RNAseq测序结果
为了确定RNAseq数据的准确性,使用qPCR在12个其他婴儿血管瘤组织进行验证。病人信息如table 1。qPCR引物如 table 2。GAPDH用于内参,使用2(-△△Ct)相对定量。

2.11 circRNAs来源基因GO,KEGG功能富集分析
基于统计模型,我们做了GO,KEGG富集分析。差异表达circRNAs来源基因用GOseq R包分析,使用基因长度进行校正。Benjamini and Hochberg’s方法对P-values进行校正。corrected P<.05的GO术语认为有显著富集。展示前10个术语。KOBAS软件分析KEGG通路,展示top20个通路。
2.12 circRNA-microRNA关系预测
miRanda (http://www.microrna.org/) 预测circRNA-microRNA关系。webserver (http://circos.ca/)绘制circRNA与miRNAs circos图。
2.13 统计分析
SPSS 20.0 software package (SPSS, Chicago, IL) an independent-sample t test 检测2组。mean±standard deviation (SD)呈现数据,并至少三重复。P<.05认为有统计学意义。
3 结果
3.1 RNAseq技术比较血管瘤和配对正常皮肤组织circRNAs表达谱
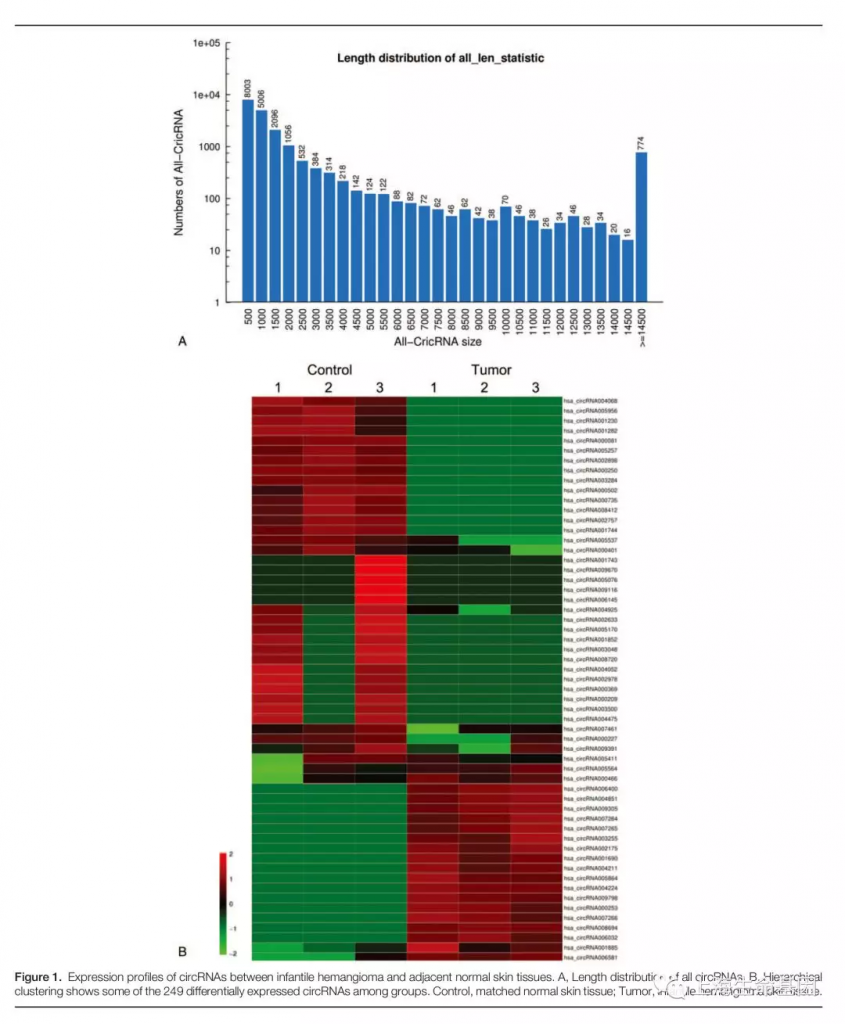
本研究收集未进行任何治疗的15个血管瘤及皮肤组织。如Table 1,病人平均年龄(标准差)是7月6天(2月15天)。为了研究血管瘤circRNAs差异表达谱,我们对3个自由选择的血管瘤组织和配对正常皮肤组织做了RNAseq。所有样品circRNAs的长度如Figure 1 A。大部分circRNAs的长度低于500nt。249个circRNAs差异表达,包括124个上调,125个下调。聚类分析展示了249差异表达的circRNAs(Fig. 1 B) (foldchange ≥ 2, P≤ .05)。
3.2 qPCR验证
为了验证RNAseq数据,我们自由选择了6个差异表达的circRNAs(table 3)。qPCR对其他12个独立的血管瘤,皮肤组织(table 1)进行定量。结果显示6个circRNAs的在qPCR与RNAseq的数据表达趋势是一致的。6个circRNAs中,hsa_circRNA001885, and hsa_circRNA006612,表达是12.33- and 7.13-fold,高于配对皮肤组织(Fig. 2)。

3.3 对差异表达的circRNAs来源基因进行GO, KEGG富集分析
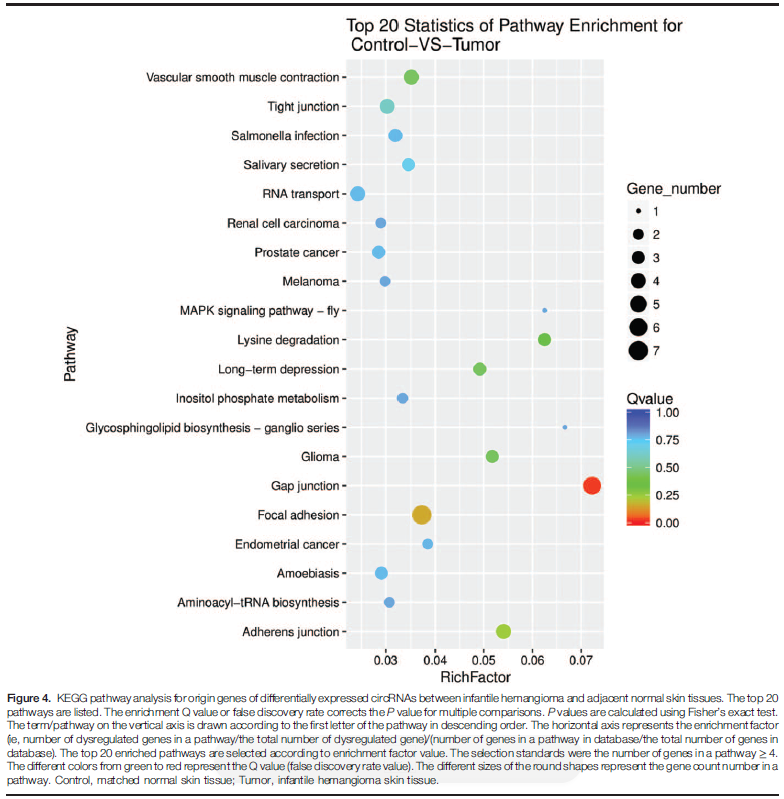
近期研究显示circRNAs可以调控来源基因的表达。因为,我们对差异表达circRNAs的来源基因进行GO,KEGG富集分析。GO结果显示来源基因大部分与cellular component organization or biogenesis, binding, protein binding, and intracellular organelle parts有关(Fig. 3)。KEGG结果显示与gap junction, focal adhesion, and adherens junction有关(Fig. 4)。

3.4 circRNA与miRNA关系
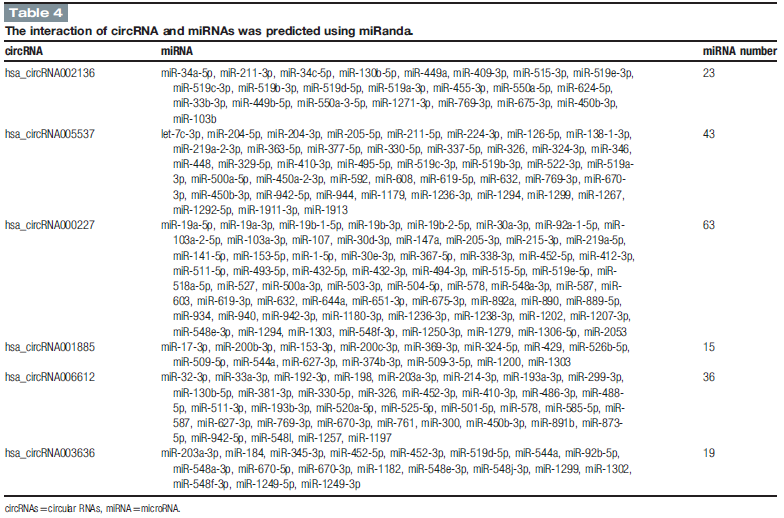
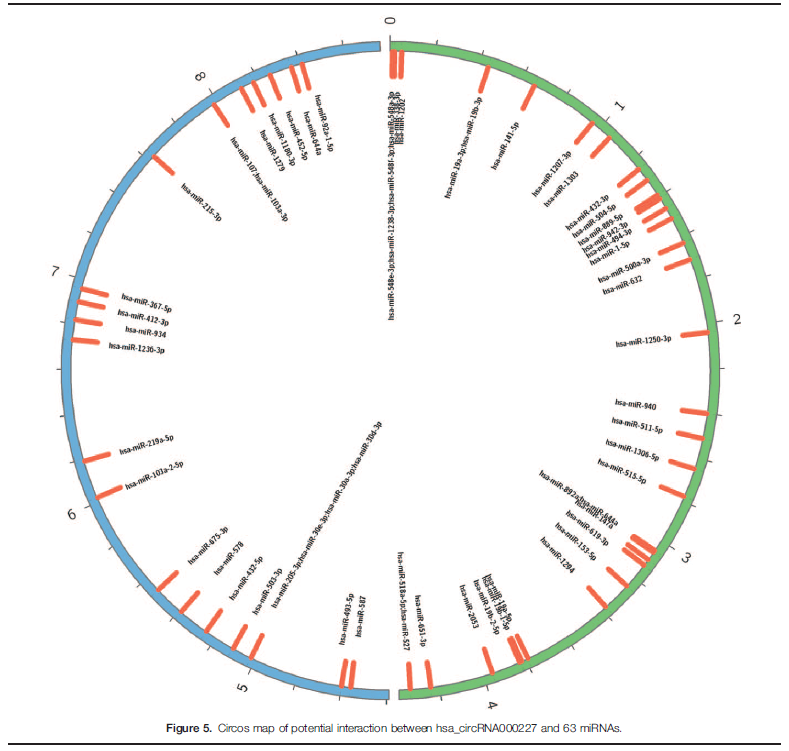
许多证据表明circRNAs可以发挥miRNA海绵吸附作用。The competitive endogenous RNAs (ceRNA)具有miRNA response elements(MREs),比如circRNAs, mRNAs, lncRNAs均能竞争结合miRNA。因为我们使用miRanda软件扫描6个circRNAs的MREs。结果显示有些miRNAs与这些特别的circRNAs有关(Table 4)。其中有63 miRNAs(高含量)能潜在结合hsa_circRNA000227 (Fig. 5), 15 miRNAs可以结合hsa_circRNA001885,36 miRNAs可以结合hsa_-circRNA006612。
4 讨论
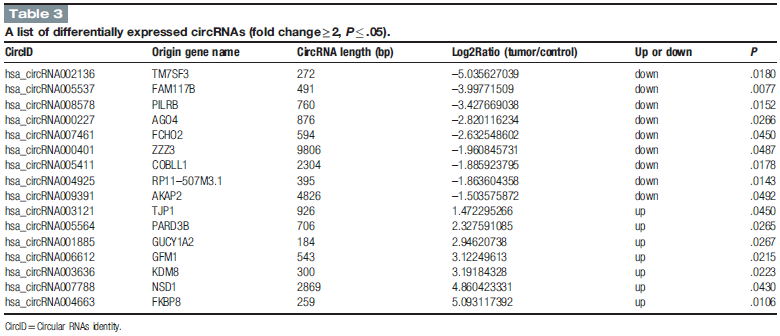
作为一种血管肿瘤,血管瘤是小孩中出现频率最高肿瘤的一种。血管瘤的致病机理得到广泛研究,并提出了几种理论。内皮驱动细胞理论,Folkman Klagsbrun胎盘理论,血管再生理论,组织缺氧理论是目前接受度最高的几种。目前为止,有报道过使用circRNA芯片研究血管瘤,并鉴定到234个上调,374个下调circRNAs。本研究,我们基于RNAseq技术,发现249个circRNAs异常表达,124个上调,125个下调。
有报道称细胞质中circRNAs的含量丰富,保守,稳定,并起源于线性RNA模板的反向剪接或者外显子跳跃。大量的文献通过RNAseq技术反向剪接发现了数以千计的circRNAs。最近有研究认为芯片鉴定circRNAs表达谱的效率要比RNAseq技术高。我们的数据与芯片的数据并不完全一致。根据来源基因,我们发现249个异常表达的circRNAs有47个来源基因可以在circRNA芯片中发现(fold change ≥ 2, P<.5)。
circRNAs最近成为一个新的明星分子在调控大量的生物学和致病过程中发挥重要作用。最近研究显示exon-intron circRNAs主要位于核内,与U1 snRNP作用通过顺式调控增强来源基因的表达量。我们的研究显示249个异常表达的circRNAs的来源基因主要与binding, protein binding, intracellular organelle part, gap junction, focal adhesion and adherens junction有关,提示这些circRNAs可能参与这些生物学过程,分子功能和信号通路的。进一步推测血管瘤circRNAs潜在的机制,有助于揭示其生物学原因并对评估和治疗血管瘤提供有用的信息。
对6个验证circRNAs(Table 3)的来源基因,我们发现TM7SF3 and GUCY1A2也能在芯片数据中找到。因此 hsa_circRNA002136 and hsa_circRNA001885可能是最有希望的研究目标。TM7SF3能通过减少内质网应激维持蛋白动态平衡促进细胞存活。过表达TM7SF3可以抑制caspase 3/7激活。FAM117B是一个新鉴定的在基因组范围内有意义的易感位点,鉴定于欧洲病例对照种群研究。作为一个AGO蛋白,AGO4能结合small RNA并介导与互补靶标RNA的分开。GFM1突变可以引起新生儿线粒体肝性脑病。JMJD5/KDM8对于哺乳动物基因组的稳定是极为重要的。因此这些特别的circRNAs可能影响血管瘤中的这些通路。需要更多的研究揭示这些circRNAs的功能。
CircRNAs可以通过碱基互补配对海绵吸附miRNA。近期有研究报道,circHIPK3能够吸附9 miRNAs,拥有18个潜在的结合位点。circMTO1能够能过吸附致癌的miRNA-9促进p21表达进而抑制肝细胞癌进程,说明circMTO1可能是肝细胞癌治疗的一个潜在靶标。我们的研究发现许多miRNAs能够结合特定的circRNAs,使其在血管瘤中发挥类似作用。一些研究报道miR-130a, miR-382, miR-9, miR-939 and the let-7家族与血管瘤的病理过程相关。因此,很有可能hsa_circRNA005537海绵吸附let-7c-3p在血管瘤进展分子机制中发挥重要作用。
CircRNAs, miRNAs, mRNAs的新关系可以为疾病的致病过程提供一个新的解释。因此,推断血管瘤中CircRNAs, miRNAs, mRNAs潜在的机制,有助于揭示其生物学原因并对评估和治疗血管瘤提供有用的信息。
5 研究思路
材料:
3个血管瘤组织,3个配对正常皮肤组织用于RNAseq,另外12个病人样品用于验证。
研究过程:
1 RNAseq建库:去除rRNA, 未去除其他线性RNA,链特异性建库, Hiseq X10 platform测序,模式为PE150。
2 使用circRNA_Finder流程鉴定和定量circRNAs。TPM数据归一化,DESeq进行差异表达分析。
3 GOseq R GO富集分析,KOBAS软件分析KEGG通路。
4 miRanda预测circRNA-miRNA关系,绘制circos图展示。
5 qPCR进行验证,GAPDH用于内参。
结果展示:
1 circRNA表达谱:病人信息(Table 1),统计circRNAs长度分布(Figure 1 A),热图展示差异表达的circRNAs(Fig. 1 B)。
2 qPCR验证:挑选6个circRNAs(table 3),qPCR定量散点图(Fig. 2)。
3 功能富集分析:GO富集柱状图(Fig. 3),KEGG富集散点图(Fig. 4)。
4 circRNA与miRNA关系:circRNAs与miRNAs结合列表(table 4),circos图(Fig. 5)。





来第一个抢占沙发评论吧!