原创作者:基迪奥生物(小师妹)
环状RNA(circRNA)可以说是近十年来科研学术界的宠儿,从种类、亚细胞定位研究到功能研究,热度有增无减。对circRNA的功能研究做得最多的是miRNA sponge作用及ceRNA效应。而随着2017年第一篇报道circRNA翻译的文章的出现,人们才发现circRNA也有可能通过翻译成多肽来行使特定的生物学功能,从此circRNA的功能研究就有了新方向(又有了发表高分文章的希望~)。
中山大学附属第一医院的张弩教授,在2018年就发表了三篇circRNA编码功能多肽的文章,分别报道了circ-FBXW7、circ-SHPRH和circPINTexon2可翻译成功能多肽,并且具有抑制胶质瘤细胞的功能[1][2][3](具体文章解读请戳 “基迪奥客户合作文章 | 翻译组测序揭示经典lncRNA形成环状RNA后可翻译功能多肽” 、“基迪奥合作客户nature子刊文章发表!”)。
这些成果发表在《Journal of the National Cancer Institute》、《Nature Communications》等高分杂志上,可谓硕果累累。文章引用次数也是很惊人,短短一年时间已经达到90了,可见该领域正在受到大家的追捧。

环状RNA编码能力分析流程,可以从RNA-seq数据中鉴定环状RNA,并通过一系列的生物信息学分析来预测环状RNA的蛋白编码能力。同时,结合翻译组ribo-seq数据,进一步找出环状RNA翻译的证据。Ribo-seq测序分析流程也是我们领先于行业的一大特色~~
下面就为大家简单介绍一下环状RNA编码能力分析流程的分析内容:
circRNA具有翻译能力需要两个基本要素:开放阅读框(ORF)和翻译启动元件(IRES)。通过生物信息分析预测circRNA是否具有ORF和IRES,可寻找潜在具有编码能力的circRNA。
1、circRNA ORF注释
由于大多数的circRNA都是来源于蛋白编码基因并且具有完整的外显子,因此我们需要预测横跨circRNA接口位置(backspliced junction)处的ORF。如果一个circRNA具有横跨junction位点的ORF,就能初步暗示其具有编码能力。我们利用cORF pipeline脚本对circRNA进行ORF注释,来寻找具有编码能力的circRNA[4]。
2、IRES预测
通常来讲,真核生物翻译只能从mRNA的5’端开始,因为翻译起始必须依赖于5’端的帽子结构。内部核糖体进入位点(Internal Ribosome Entry Site,IRES)则是一段核酸序列,能够使蛋白质翻译起始不依赖于5’帽子结构,而是直接从mRNA内部开始翻译。由于circRNA不含有5’帽子,因此其翻译必须要依赖这些翻译调控序列。
我们利用IRES finder进行circRNA的IRES预测[5]。IRESfinder是一个用于识别真核细胞内RNA内部核糖体进入位点的python包,可以用于进一步搜索核心IRES区域。预测结果会给出打分最高的一段区域,该区域可以认为是该circRNA的IRES序列。
3、翻译分析
以上两步只是从生物信息学的角度去预测circRNA的潜在编码能力,那么有没有什么数据能够证明该circRNA真的能编码呢?翻译组数据就是一个很有力的证明。翻译组测序是指对与核糖体结合的正在翻译的RNA片段进行测序。从ribo-seq数据中寻找支持circRNA翻译的reads,可以更有力更可靠地说明该circRNA潜在可翻译。具体做法是将比对不上线性RNA的reads去比对circRNA的junction位点,找出并统计支持circRNA翻译的reads。
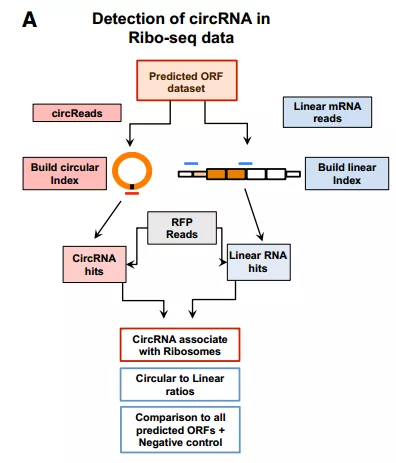
综合以上,利用环状RNA编码能力分析流程进行环状RNA编码能力的预测,包括了以下三方面:
(1)对circRNA的ORF进行注释;
(2)利用IRESfinder软件预测circRNA的IRES序列;
(3)若同时有ribo-seq数据,可将比对不上线性RNA的reads去比对circRNA的junction位点,找出并统计支持circRNA翻译的reads。
当然,要发表高分文章,做完以上三步还是不够的,还需要进一步通过质谱技术鉴定该circRNA编码的多肽,并且进行多肽的分子细胞功能、机制研究。具体的研究思路可以参考上述提到的张弩教授的circPINTexon2编码多肽文章。研究新领域、并且具有完整的研究逻辑和充分的证据,这才是发表高分文章的必要组成要素。
参考文献
[1] Yang Y, Gao X, Zhang M, et al. Novelrole of FBXW7 circular RNA in repressing glioma tumorigenesis[J]. JNCI: Journalof the National Cancer Institute, 2018, 110(3).
[2] Zhang M, Huang N, Yang X, et al. Anovelprotein encoded by the circular form of the SHPRH gene suppresses gliomatumorigenesis[J]. Oncogene, 2018, 37(13): 1805.
[3] Zhang M, Zhao K, Xu X, et al. A peptideencoded by circular form of LINC-PINT suppresses oncogenic transcriptionalelongation in glioblastoma[J]. Nature Communications, 2018, 9(1).
[4] Pamudurti N R, Bartok O, Jens M, et al.Translation of circRNAs[J]. Molecular cell, 2017, 66(1): 9-21. e7.
[5] Zhao J, Wu J, Xu T, et al. IRESfinder:Identifying RNA internal ribosome entry site in eukaryotic cell using framedk-mer features[J]. Journal of genetics and genomics= Yi chuan xue bao, 2018,45(7): 403.





来第一个抢占沙发评论吧!