上一期我们的武器库里又增添了一把利器,通过统计模型获得高特异性、敏感度的circRNA预测结果 KNIFE,支持有参及 de novo 比对识别,多见于人类研究中。今天的推送将给大家带来一个基础的数据库 circBase 逐渐壮大的过程。
愿景
作者拟建数据库最原始的目有以下几点的:
1、每一个circRNA应该有据可循,能够被归类以及访问;
2、circRNA应该包含基因组信息,以及可用的表达或者调控数据;
3、搜索框应该接受多种形式的词条,比如序列、基因或基因区间等;
4、提供交互式多种数据形式的下载内容;
5、用户能够上传自己的表达数据并且实现可交互。
因此,这个数据库还是灰常良心和稳定的,自2014年发布以后,一直有人在投入维护,并且在行业内形成一个良性循环,诸如提供circRNA的命名规则,提供 UCSC gemone browser信息等。
功能介绍
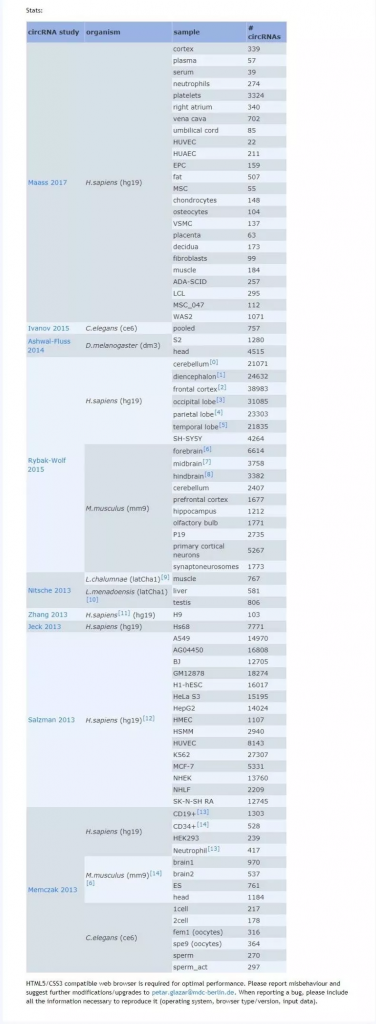
首先我们来看数据库上涵盖的数据信息,截至发稿,主要包括的测序物种有 Homo sapiens, Mus musculus, Caenorhabditis elegans, Latimeria等。
circBase 获取数据主要有以下三种方式:
simple search
List search
Table browser
涵盖绝大多数简易搜索的词条,包括输入DNA or RNA序列后,使用 Blat获取反向互补序列或者获得包含 circular junctions的circRNA序列;符合规则的输入基因组位置能够获得circRNA overlapping repeats等一系列信息;当然也可以通过简单的关键词搜索,获得标准格式的 12-column BED 文件。简单的搜索内容如下图:
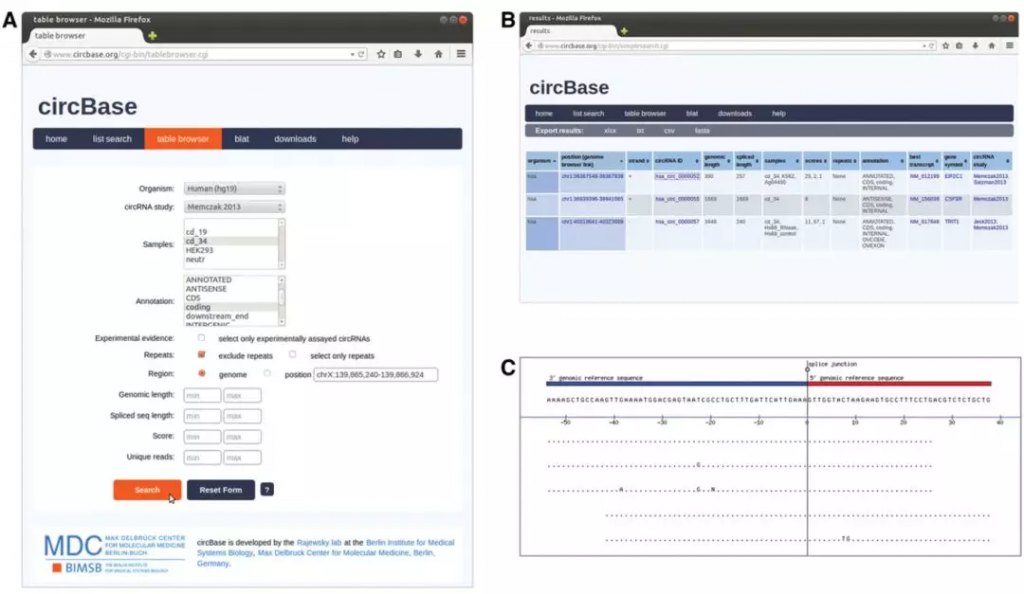
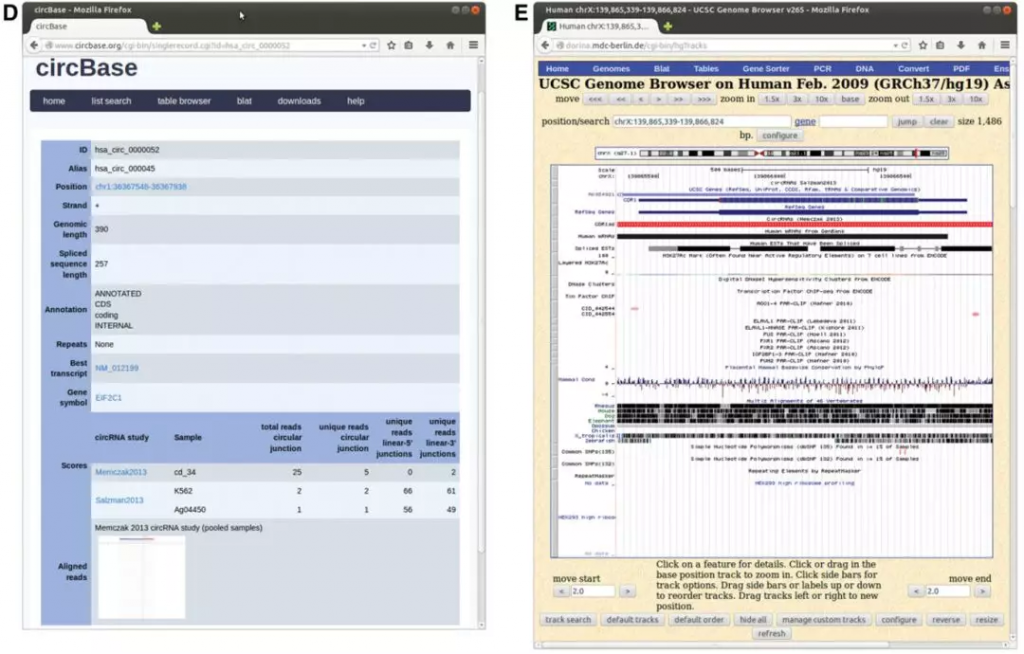
下载数据格式包括 .xlsx, tab-separated .txt 以及FASTA format,FASTA文件通常以 tar.gz打包。在数据库上,作者非常友好的将如何发现circRNA所需要的所有代码 (其实就是find_circ),以及用于测试的自测RNA-seq 数据放入 downloads 页面,详细内容可见 README。
注意事项
关于数据标签的展现形式,比如circBase上 hg19 for H. sapiens, mm9 for M. musculus and ce6 for C。因此可以通过 liftOver 上进行转换。有需求上传的用户,可以直接与作者联系。
这是数据库系列的第一篇简单推文,后续还会提供结合工具不同方向内容的数据库,目前已有不少实验室直接从数据库上获得关键序列进行后续验证而发高分文章,值得关注。





来第一个抢占沙发评论吧!