转自:生信菜鸟团
上一期我们给大家带来一个基础的数据库 circBase 逐渐壮大的过程,集人类、小鼠、线虫等模式动物的circRNA研究进展,并支持上传下载,提供 UCSC 基因组信息查询。目前,已有多篇文献基于此进行下游验证研究,并发表文章。例如:

或者,

今天的推送将介绍一个全球高访问热度的人类癌症特异 circRNA 数据库 cancer-specific circRNA database (CSCD, http://gb.whu.edu.cn/CSCD)。那个白点其实就是我正在访问的位置:)
数据库最新概况
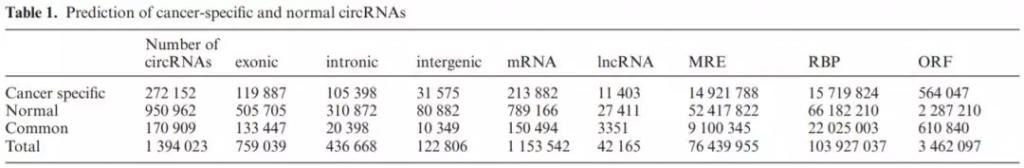
CSCD的数据包含87个癌细胞系样本、总共228个RNA测序数据集(total RNA with rRNA depleted or polyA- enriched),通过4种常用的circRNA识别算法综合分析,总共呈现272,152条癌症特性性circRNA,其中950,962条circRNA为癌症特性性表达。
愿景: CSCD提供一下几个角度,拟促进circRNA在癌症的研究:
癌症特异circRNA (Cancer-specific circRNAs, CS-circRNAs)研究;
1、CS-circRNAs的miRNA sponge 功能;
2、CS-circRNAs与蛋白 (RBP) 互作;
3、CS-circRNAs的翻译 (ORF);
4、CS-circRNAs的可变性剪切。
功能介绍
CSDS数据库的搜索界面主要能够通过以下几种词条进行癌症特异性circRNA信息搜索:
1、细胞类型 ( cell type)
2、基因标签 ( gene symbol)
3、亚细胞位置 ( cellular location)
在输出列表中,用户将获得每一种癌症相关circRNA的所有信息,包括样本来源、circRNA来源基因的信息,基因组坐标位置、注释,Junction Reads的数量,circRNA 与线性基因之间的比率,外显子剪切的信息,circbase id,细胞位置,获得circRNA所预测使用的算法以及表达丰度。并提供下载功能。

亮点介绍
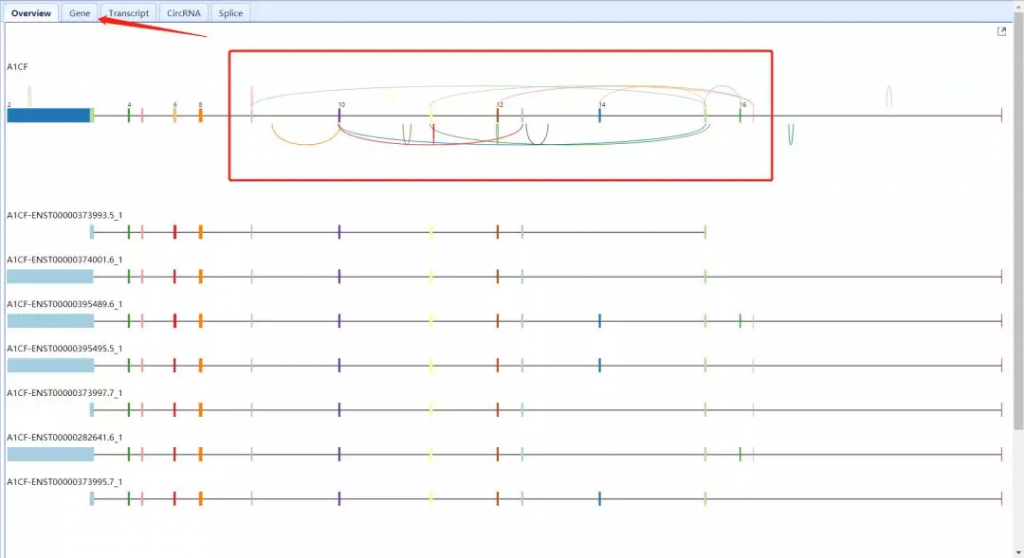
单击 Gene symbol 或者circRNA id将在有边框中获得一系列可视化的材料。点击 UCSC将进一步获得该片段在染色体上的具体信息。在右上面板中,以圆弧连接预测 donor 及accepted site的形式来表现circRNA的形成种类,在上方的标签中进一步展示circRNA来源基因的注释以及接头reads的数量、位置等信息。
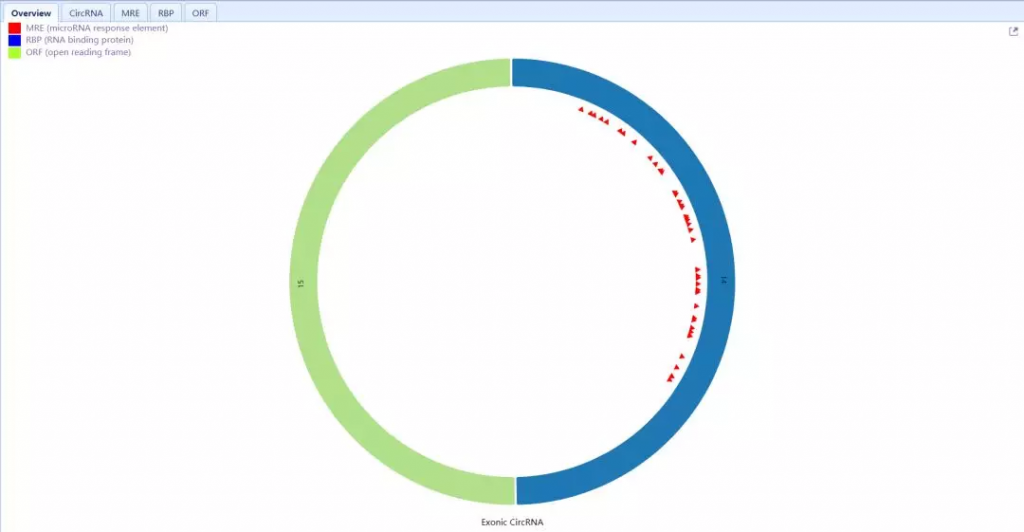
在右下面板中,以不同颜色代表目标circRNA序列上,预测的 MRE, RBP, ORF信息。在标签中能够获得更详细的内容,包括MRE位点的类型、起始坐标以及miRNA家族信息;RBP的名称、坐标位置;以及ORF的位置信息。
注意事项:
目前,CSDS支持 GRCH37及GRCH38两个版本的人类基因组信息,因此在使用前自行选择。
小结:
总体来说,虽然数据库整体以整合现有数据为主,提供多角度的cs-circRNAs信息。界面比较友好,访问速度也快,链接按钮都可用,对于设计课题初期的相关癌症样本circRNA研究信息获取具有极大帮助。
部分参考文献
Tang, Weiwei, et al. "CircRNA microarray profiling identifies a novel circulating biomarker for detection of gastric cancer." Molecular cancer 17.1 (2018): 137.
Chen, Yajun, et al. "Circular RNA circAGO2 drives cancer progression through facilitating HuR-repressed functions of AGO2-miRNA complexes." Cell Death & Differentiation(2018): 1.
Xia, Siyu, et al. "CSCD: a database for cancer-specific circular RNAs." Nucleic acids research 46.D1 (2017): D925-D929.





来第一个抢占沙发评论吧!