转自: 生信菜鸟团(微信公众号)
上回我们说道,由于能够有针对的设计特异性microarry 探针,因此能够接近100%获得circRNA探测信号,并且在低丰度circRNA的捕获上以及抵抗反向剪接所带来的噪音干扰上有比较好的表现。因此在文章作者讨论的组织样本中,microarry会是一个比较好的选择,也为后续circRNA作为新型诊断指标提供了较好的研究原材料。而RNA-seq方法获取circRNA数据,还有没有提升的空间么?同样,我们来看一篇文章。

背景介绍
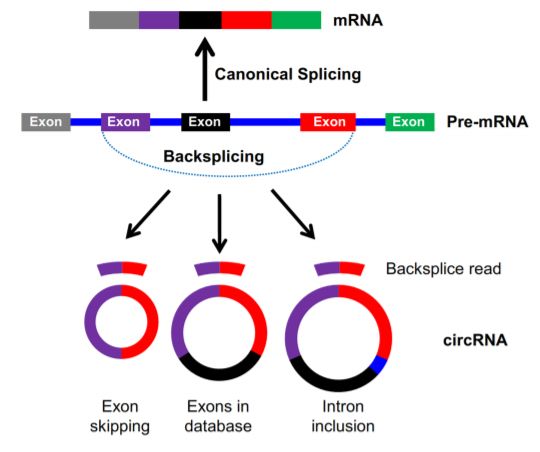
凭借高通量RNA-seq测序分析,能获得任何基因组区域上长度范围在几十nt到几kb的转录产物,其中包括大量的circRNAs。根据来源序列,绝大多数的circRNAs来源于编码RNA的外显子;而来源于内含子序列的circRNAs称作circular intronic RNAs (ciRNAs);也包括保留了外显子间内含子序列的exon–intron circRNAs, or EIcircRNAs。
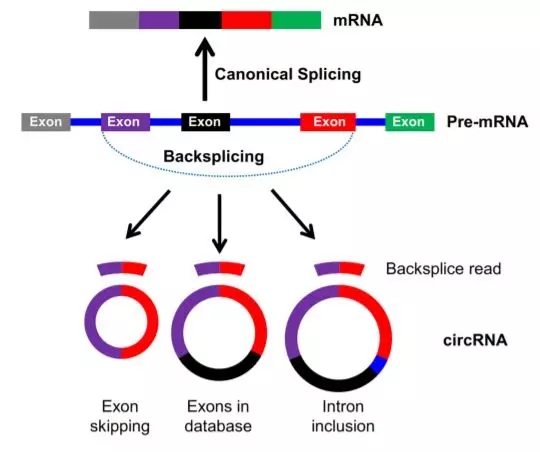
研究过程
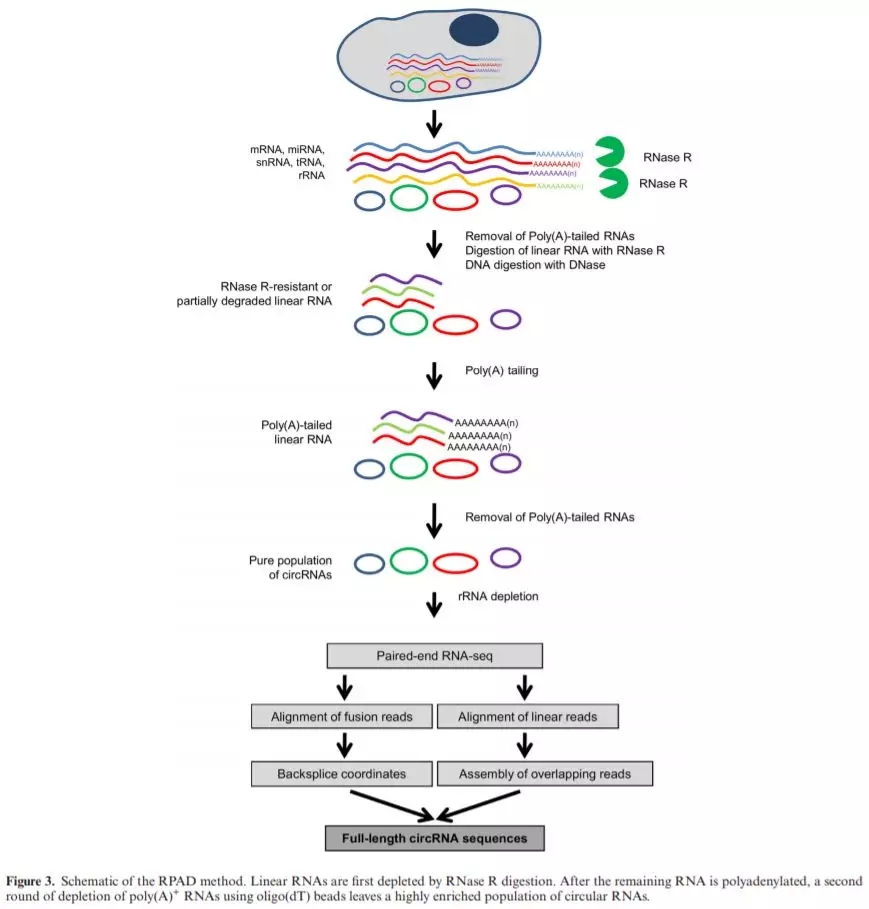
由于circRNA特殊的拓扑结果,故在构建测序文库时会采用核酸外切酶能够有效地富集环状RNA,但是由于线性RNA二级结构的存在,这种方法并不能完全去除所有的线性RNA。而剩余的线性RNA的表达水平可能也远远高过于同源的circRNA,同样也会阻碍我们通过qRT-PCR或者Northern blot的实验方法对circRNAs的定量和定性分析。因此,作者在文章中提出一个试验方法:RPAD (RPAD: RNase R treatment followed by Polyadenylation and poly(A)+ RNA Depletion),并验证该方法能够几乎完全去除线性RNA的干扰。
RPAD大致的方法如下:首先使用RNase R去除线性RNA,然后对 '幸存' 的RNA 3’游离羟基(-OH)多聚腺苷酸化 (polyadenylating)并随后消除这些含有poly(A)尾的RNA。而作者使用这种方法制备的RNA样本测序后,分别在宫颈癌HeLa细胞和C2C12成肌细胞中(cervical carcinoma HeLa cells and C2C12 myoblasts),鉴定获得了很多新的circRNAs序列。RPAD方法的示意图如下:
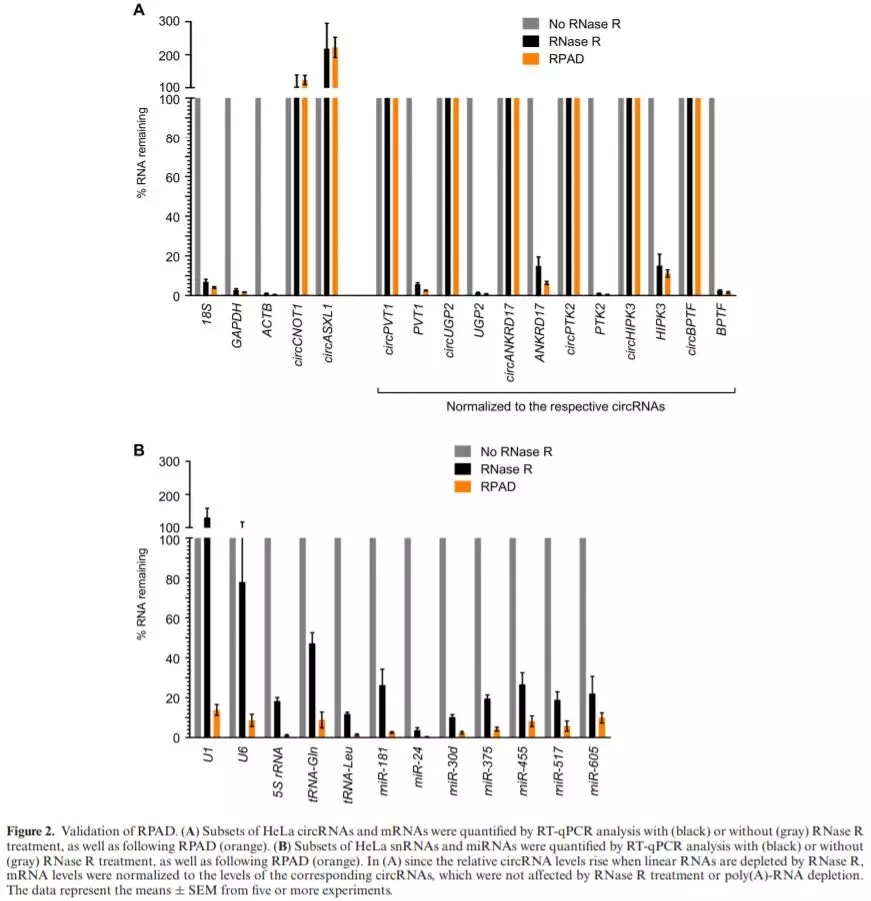
根据作者的结果,我们能够看到在高浓度的RNase R及长时间的处理时间下,仍有2-20%mRNA未被消化(Figure 2A),而由于双链RNA及广泛存在的二级结构也干扰RNase R的消化。且RNase R处理不能降解snRNA U1和U6,而其他测试的small RNAs 中,2-30%的序列保持完整(Figure 2B)。而通过RPAD方法后处理,ANKRD17 mRNA从15%将至7%(Figure 2A),而miRNA和snRNA极度减少(Figure 2B)。
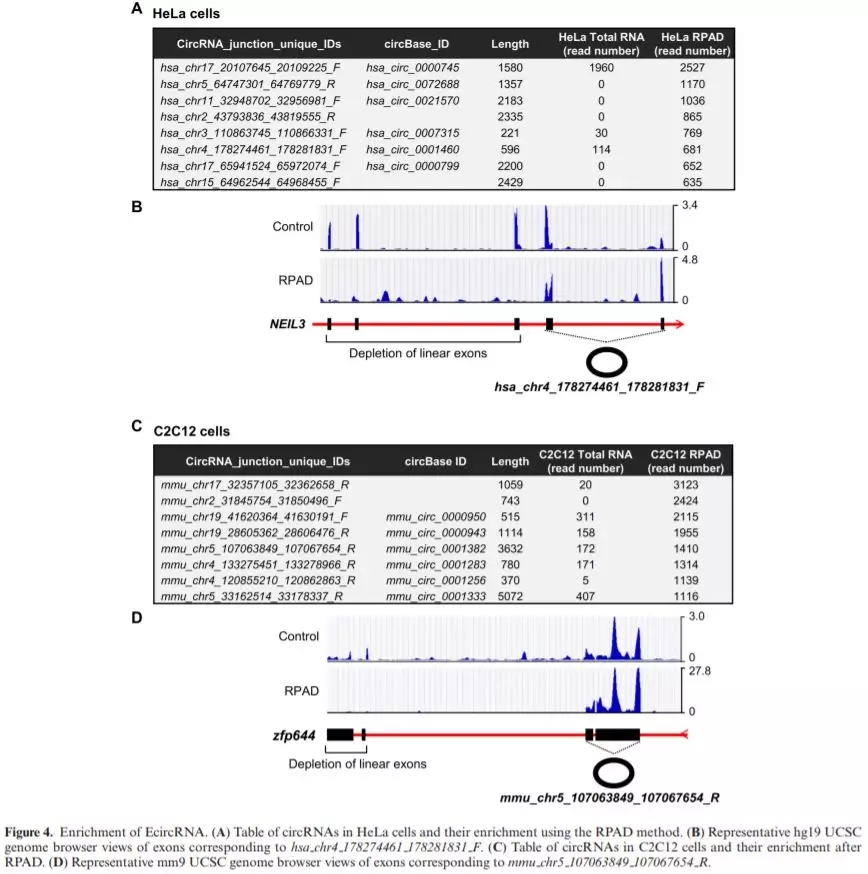
随后作者使用RPAD方法后获得的数据,对鉴定的circRNA序列的组成进行讨论。相比较普通RNase R处理后的测序分析结果,RPAD方法在circRNA全长鉴定、新鉴定序列 (基于circBase)特别是在IcircRNA的鉴定上有较好的表现(Figure 4)。
小结
从实验结果来看,RPAD方法能够分类获得高纯度的circRNA,使高通量RNA-seq更加全面鉴定circRNAs,从而进一步丰富阐释circRNA的序列、表达丰度及功能。例如通过CircInteractome互作工具能够进一步预测circRNA与RBP相互作用,展示在细胞生理学上发挥的功能。同时,新鉴定的IcircRNA序列,能够校准CLIP数据库中对内含子的注释。当然更加全观的数据,匹配合适的鉴定算法将会大力推进circRNA的研究。




来第一个抢占沙发评论吧!