转自:生信菜鸟团(微信公众号)
编者按
circRNA以其独特的拓扑结构,常占生命过程中重要角色。
也正因为其独特的环形序列,circRNA与常规的RNA实验流程也存在差异。
今天就整理一下实验技术创新研究成果。
前景提要
首先我们来回顾一下以往推送中提到的方法。根据不同的富集方法,可以将高通量数据获得分为芯片及 RNA-seq。microarry 方法中能够针对反向可变剪切位点设计特异位点富集 circRNA,因此能够接近100%获得 circRNA 探测信号,并且在低丰度 circRNA 的捕获上以及抵抗反向剪接所带来的噪音干扰上有比较好的表现,但是由于特异探针设计可能带来敏感度及成本问题。而便捷、常见的方法使用的是 RNA-seq。
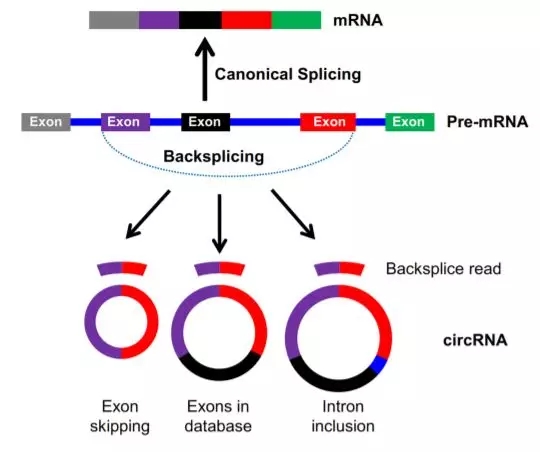
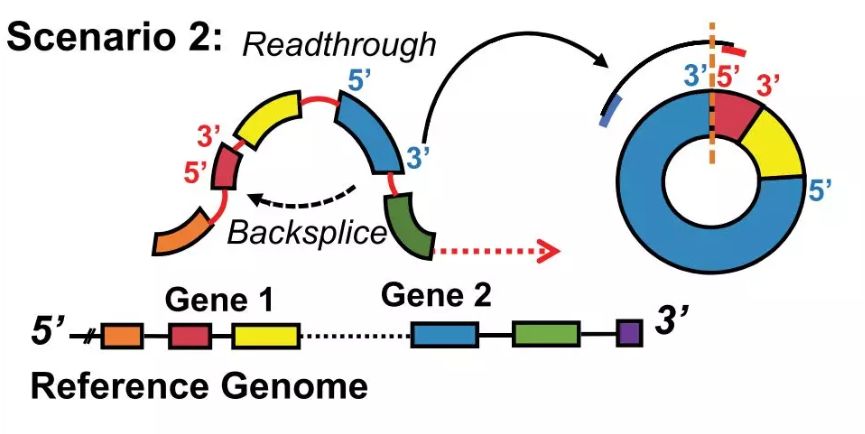
常规的 RNA-seq 满足任何基因组区域上长度范围在几十nt到几kb的转录产物的要求,其中当然包括 [无处不在的] circRNAs。根据来源序列,简单地能够将 circRNA 分为来源于编码RNA的外显子 circRNA;内含子来源的 ciRNAs 及 保留了外显子间内含子序列的 EIcircRNAs。最近的cell报道了一个新分类的 rt-circRNA,这是一类由融合基因临近的两个外显子组成的一类新型 circRNA,模型如下图所示。

根据优化的步骤不同,现有 circRNA 文库构建及检测手段优化有一下几种:
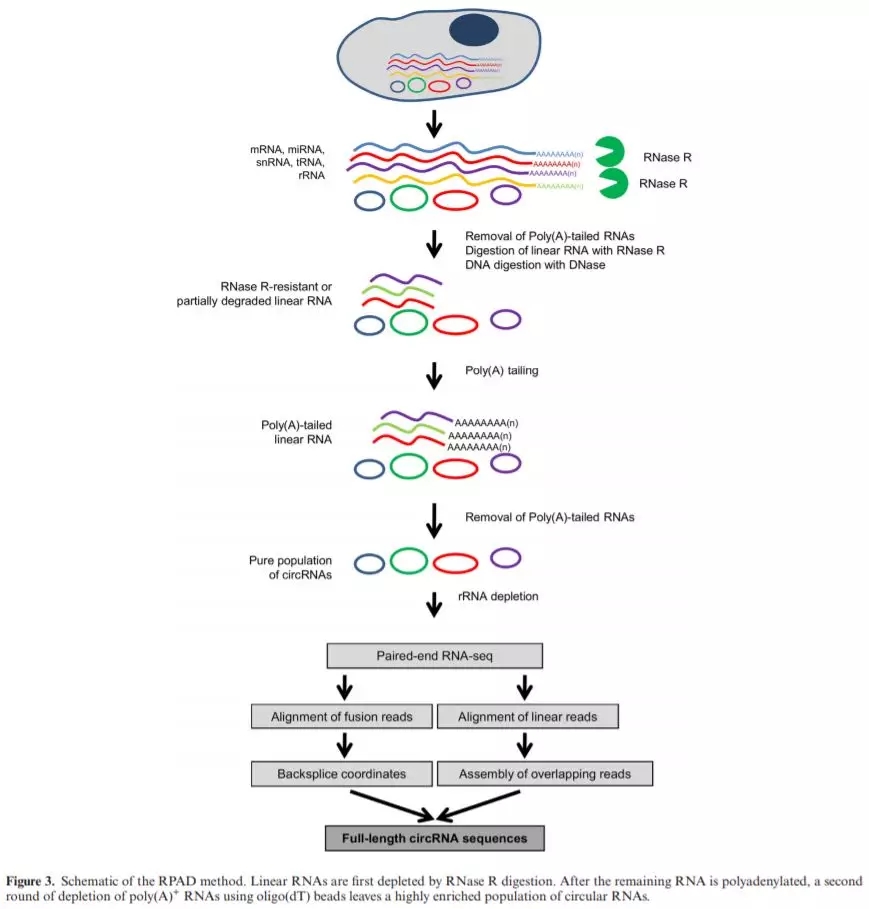
建库优化 [RPAD]
目前构建 circRNA 特异文库的方法一般先去 核糖体RNA 后会使用 RNase R 再去除线性RNA构建文库。[一种提升circRNA-seq数据全面性的方法] 作者在文中提出的 RPAD (RPAD: RNase R treatment followed by Polyadenylation and poly(A)+ RNA Depletion) 方法能够对然后对 [幸存] 的RNA 3’游离羟基(-OH)多聚腺苷酸化 (polyadenylating) 后再次消除这些含有 poly(A)尾的RNA,能够几乎完全去除线性RNA 的干扰。示意图如下:
新的测序方法 [exome capture sequencing]
该方法被应用在19年2月的 Cell 期刊上[一个新的circRNA测序建库方法介绍],VCaP 前列腺癌细胞测试中,外显子捕获方法测序能够获得大多数Ribo-Zero文库中circRNA (Fig B),且保留了 circular-to-linear fractions (Fig C)。而后经RNase R消化后的结果 (Fig D) 也证实外显子捕获文库构建的方法获得 circRNA 为真实存在。

相较于oligo(dT)磁珠富集mRNA来说,exome-capture (short “capture”) RNA-seq library preparation 有如下的特殊的方法:
Unique to capture transcriptomes is an overnight capture reaction (RNA-DNA hybridization) using exon-targeting RNA probes, followed by a washing step, and an additional set of PCR cycles.
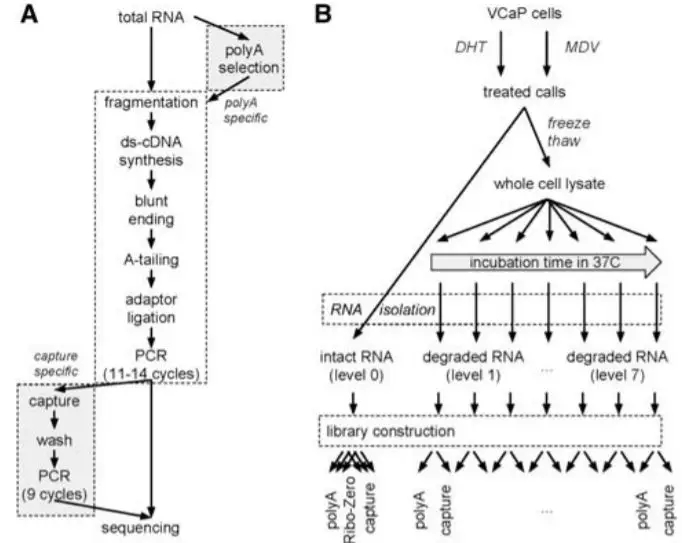
该方法能够精确且无偏好地评估 RNA丰度,转录本覆盖度。构建文库所需的 总RNA 起始浓度为0.1-3ug (100ng样本需要提高PCR循环数)。
定量实验
- 绝对定量
上上期推送中 [PP&PJ研究思路介绍] 植物作者根据 junction read 设计引物后,使用dPCR进行绝对定量。dPCR 的优势在高灵敏度,能够获得低丰度的 circRNA 表达。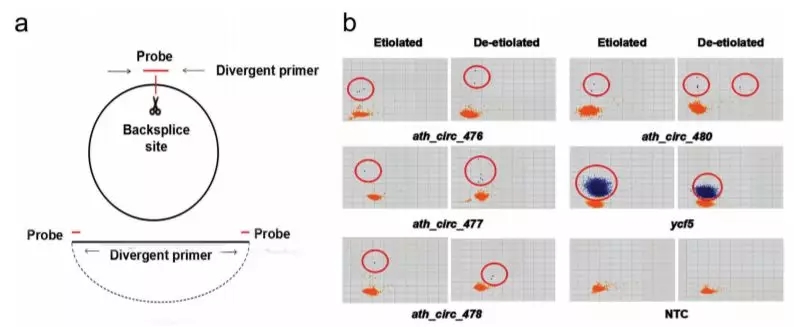
- Nanostring™nCounter®技术
基于 colour-coded DNA 探针与细胞RNA杂交技术结合数字计数,作者在在新鲜冷冻组织和福尔马林浸泡固定在石蜡上的样品中检测到52个唯一的 circRNA 序列。由于 DNA探针 的杂交直接在 RNA 而不是cDNA 上进行,因此该方法解决了定量过程中的PCR偏好性问题。但此方法成本很高。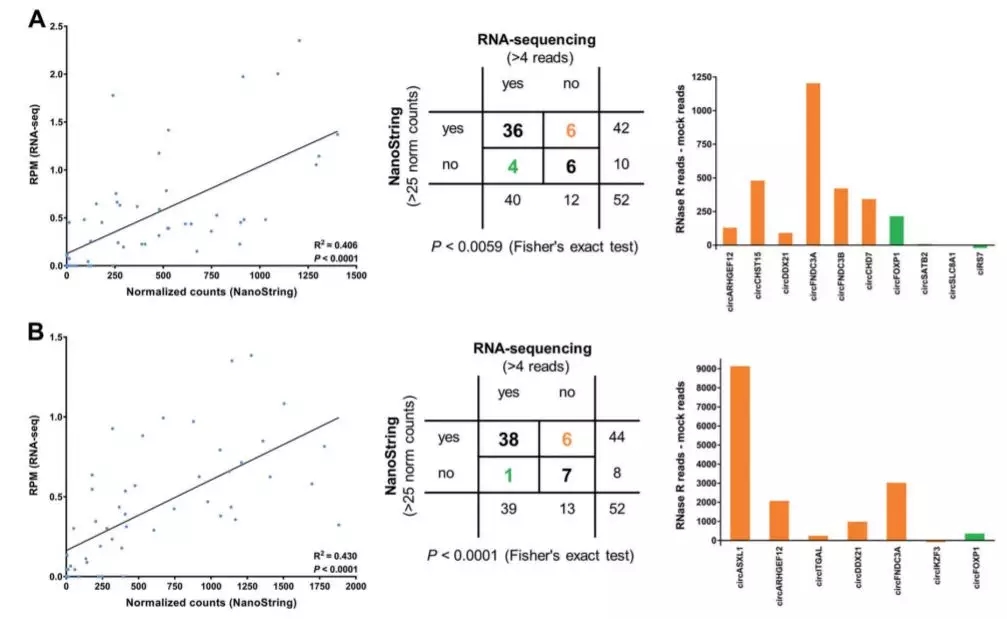
Fig. Comparison of circRNA expression data by NanoString and RNA-seq for the cell lines where RNase R experiments were performed
- SplintQuant
这是一篇最新的文献报道。在此方法中,一种经过修饰的 PBCV-1 DNA连接酶 ,能够连接相邻的 单链DNA寡核苷酸。通过将 DNA寡核苷酸 靶向直接位于circRNA 背离接头侧翼,我们可以检测特异性 circRNA,其不受同源线性 RNA、含内含子序列的 circRNA 及目标 circRNA 的片段大小。
将 M13 通用引物序列整合到 circRNA 特异性寡核苷酸的侧翼上,就能在没有逆转录(qPCR)的情况下对连接的 DNA探针 进行定量PCR,由此准确定量甚至低丰度的 circRNA 转录物。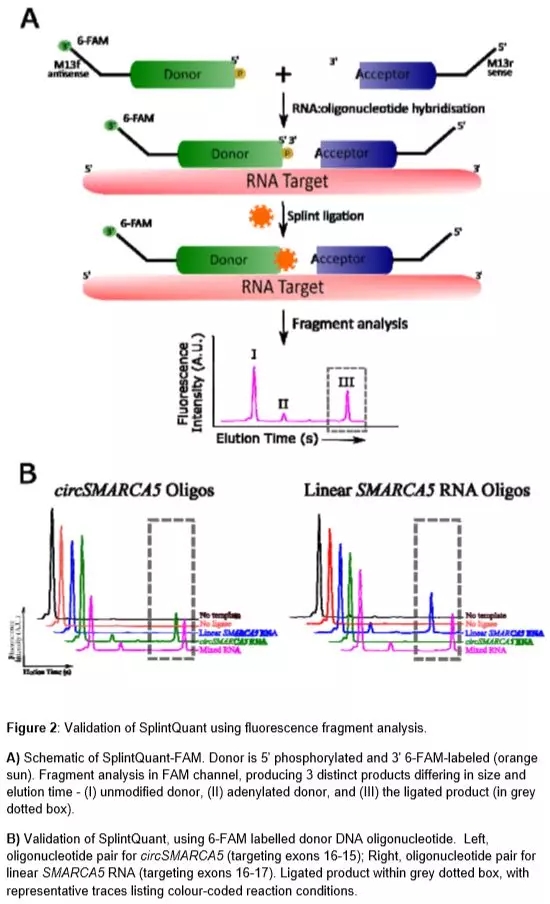
小结
19年单在 Cell 已刊登五篇大作 [Cell 文献笔记⑤],足以说明 circRNA 已成为科研高频热点。除了有生物学意义的实验设计是成功的一半外,实验方法和验证手段也显得非常重要。除了常规的荧光定量和Northern-blot 验证外,最基本半定量PCR 加由 Sanger测序 的方法依旧是简单有效的检验手段。当然,上述的几种优化 circRNA 数据获取、定量的方法也值得参考,希望对你有所帮助。



来第一个抢占沙发评论吧!