|
前言
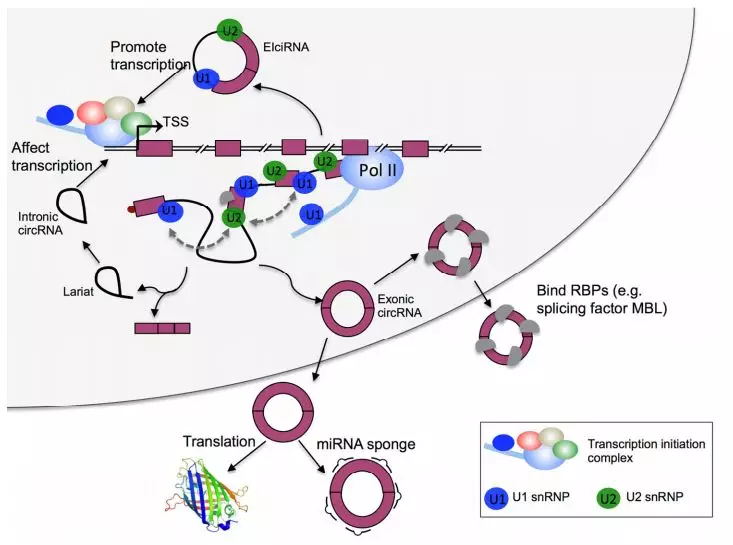
环状RNA的功能自从2012年高等动物体内被发现和报道后,其功能的研究就一直是热点。环状RNA功能与亚细胞定位相关。
(1)细胞核:可以参与转录调控,例如EIciRNA。
(2)细胞质:miRNA Sponge作用以及对应产生的ceRNA效应,是最常见的研究套路。环状RNA的miRNA sponge作用,其风
向标来自于最早的两篇Nature文章[2,3] 。这两篇文章都报道环状RNA CDR1as对miRNA miR-7的sponge作用,从而引发了环状RNA的miRNA sponge研究的热潮。但实际上,环状RNA的sponge作用可能被高估了。因为CDR1as是环状RNA中极其罕见的特例:
a) CDR1as 表达丰度非常高(在所有环状RNA中排第一),高到甚至与内参基因相当。
b) CDR1as 上有高达73个miR-7的结合位点,这在环状RNA中也是非常罕见的特例。
对于绝大部分环状RNA表达丰度非常低,且不一定包含如此高密度的miRNA结合位点,所以大部分环状RNA其对miRNA吸附能力是有限的。因此如果对丰度不高的环状RNA开展sponge研究,在细胞或动物水平的效应可能不会十分显著。
随着2017年的第一篇报道环状RNA被发现可翻译[3], 环状RNA翻译为环状RNA的功能研究提供了新的方向。从逻辑上说,多肽属于微量就可以起到巨大效应的分子,所以对于较低丰度的环状RNA更有可能通过翻译多肽来起到生物学作用。
中山大学附属第一医院的张弩教授(基迪奥合作客户)在之前的系列研究中,分别报道了蛋白编码基因(FBXW7和SHPRH)在形成环状RNA后形成新的功能多肽,且具有肿瘤抑制功能,相关结果分别发表在JNCI和Oncogene[4,5]。
在2018年,他利用翻译组数据又挖掘到非编码RNA linc-PINT形成环状RNA后可被翻译形成功能多肽,进而抑制多种癌基因的转录延伸,抑制恶性胶质瘤的发生和进展,相关结果发表在Nature Communication[6]
近期,张弩教授团队利用上一篇NC文章的翻译组数据,挖掘到的另外一个可以翻译多肽的环状RNA circ- AKT3,证明circ-AKT3 可以翻译一个174个氨基酸的多肽。体内外研究显示AKT3-174aa蛋白通过竞争性结合,调控激酶PDK1的效应,从而负调控PI3K/AKT信号通路,抑制脑肿瘤的发生发展,相关结果近期在Molecular Cancer(IF=10.68)在线发表。
至此,张弩教授团队已经有4篇环状RNA翻译多肽的高分文章发表,约占据全世界该细分领域高分文章的一半。而基迪奥公司基于已有的翻译组数据也表明,环状RNA翻译可能是非常普遍的现象(一组翻译组数据,往往可以挖掘到成百上千潜在可以翻译的环状RNA)。
当然,随着报道不断增加,环状可翻译这个小众领域慢慢被大家接受。未来,我们如果研究circRNA的sponge功能,可能要面临审稿人的追问:你怎么证明它的功能仅仅是因为sponge,而不是来自翻译多肽?
基迪奥生物为该项目提供了建库、测序和生物信息服务,通过高质量的专业服务助力合作客户实现持续的高分文章产出。对翻译组技术感兴趣的老师同学,也可以直接联系基迪奥各地的销售。
图2 文章截图(IF=10.68)
文章概要
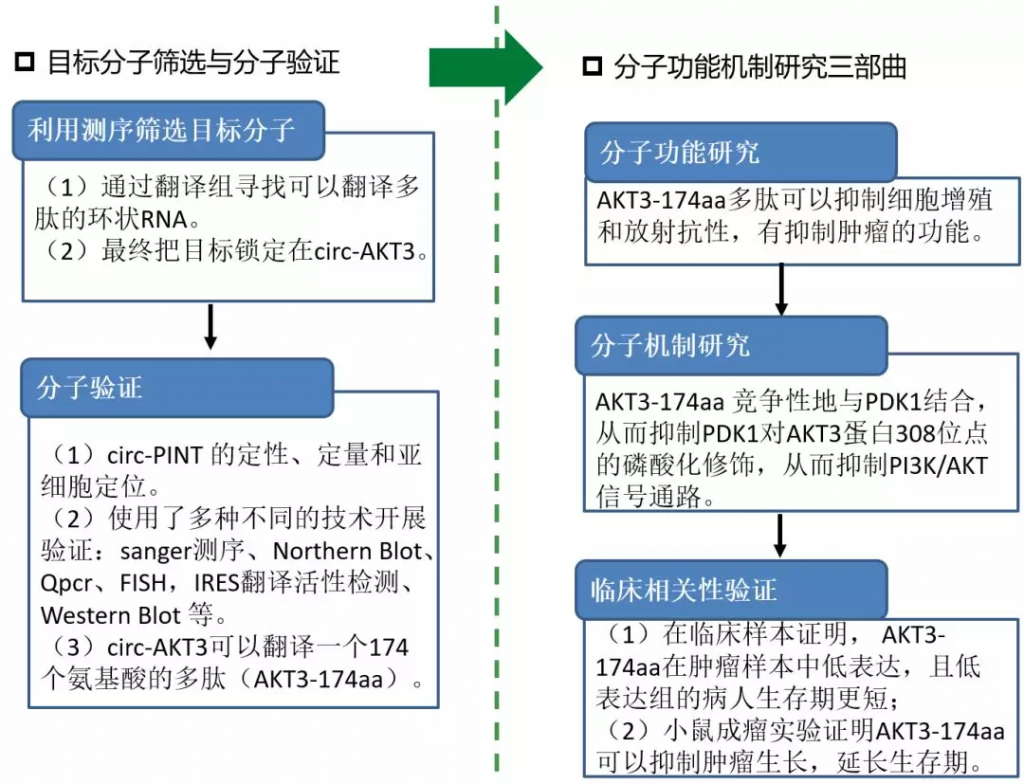
这篇文章的主要思路:
(1)从翻译组测序结果中,锁定了一个明星基因AKT3的环状剪切形式hsa_circ_0017250(环状RNA本质上就是基因的一种特殊可变剪切形式),其潜在可以编码174个氨基酸的多肽。
(2)多种手段证明circ-AKT3可以翻译多肽。
(3)医学功能分子研究三部曲:分子功能、分子机制、临床相关性研究,都证明了AKT3-174aa可以翻译多肽,这个多肽有抑制肿瘤细胞的作用。
图 3 该文章的研究思路概要
经验分享
如果翻看这篇文章,可以发现这篇文章的结构与上一篇NC文章高度相似。这类文章的总体思路:利用组学找到一个有创新性的分子,套上经典的医学临床功能机制研究(三部曲),就可以达到较好的文章产出。这里主要补充两点之前没有提过的问题:
(1)关于来源环状RNA的多肽的western验证
环状RNA可翻译是一个相对较新的方向。新的事物总是会不断受到质疑。所以,这个领域目前的高分文章,为了证明预测的多肽存在且来源环状RNA,作者总是要用多个不同的方法证明。不过,随着这个领域慢慢文章多起来,审稿人可能就慢慢不会那么苛刻。在诸多证明环状RNA可以翻译的方法中,western blot是最铁的证据之一。当然,最可信的抗体是找环状RNA多肽的特异序列的对应抗体。
这里有些背景知识:
a)环状RNA虽然是只使用的母基因部分的外显子,可以理解为是一个截断的RNA。但已有的报道发现,环状RNA编码的多肽,通常起始密码子和母基因的起始密码子在同一个读码方式里,只是起始位置不同而已。再细一点解释,一段核酸序列在同一个方向上编码,理论上有三种编码模式,但环状RNA通常不会因为翻译起始位点延后而发生移码突变,而是使用母基因读码框中的另外一个ATG作为起始。
b)环状RNA以绕圈的方式翻译。由于其长度常常不是3的倍数,所以完成第一圈翻译后,第二圈自然就会产生移码突变,从而在多肽的C端(蛋白翻译是从N端到C端)产生母基因无法编码的特异性肽段。
一句话概况就是:环状RNA的读码框具有N端的保守性和C端的高可变异性。
因此,如果环状RNA翻译超过一圈(既第一圈没有遇到终止密码子),我们潜在可以利用特异性抗体来区分多肽是来源环状RNA还是母基因。
但这个策略有两个问题:
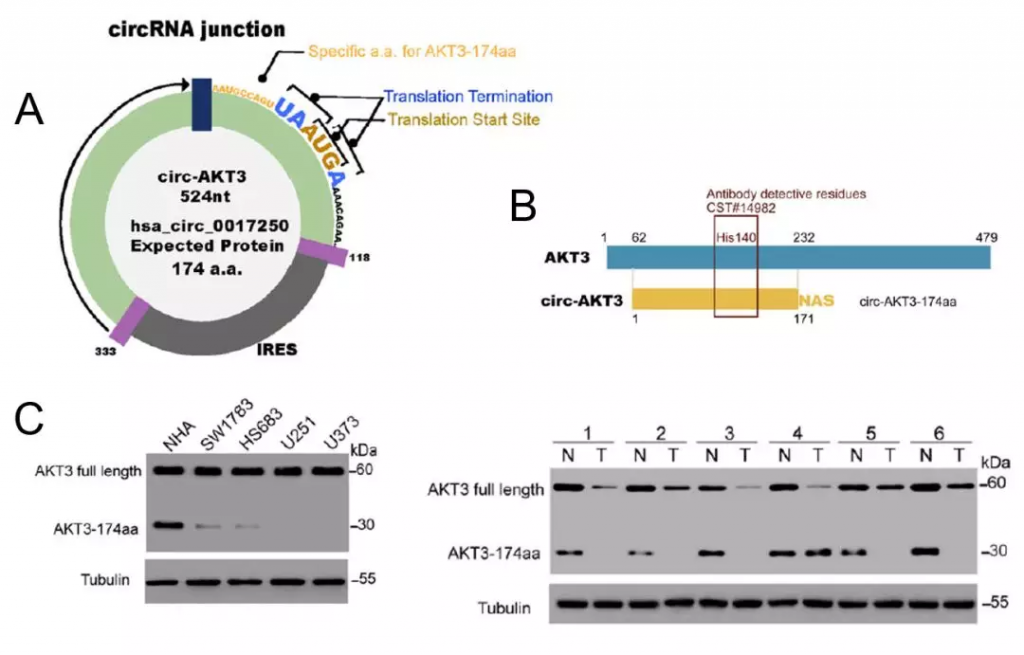
a)环状RNA有可能翻译不足一圈,或者虽然过了一圈但特异性的肽段C端还很短就遇到终止密码子了。例如,AKT3-174aa特异的部分仅仅1个氨基酸(图4A)。如果特异性部分少于8个氨基酸,就很难保证抗体特异性。
b)定制特异性抗体的周期和费用都很高。
这时候,还有一个退而其次的方法,看看是否可以找到合适的针对母基因蛋白的商业化抗体。如果商业化抗体识别的多肽序列,也包含在环状RNA多肽序列中,就可以用western blot识别和区分全长蛋白和环状RNA多肽。例如,AKT3母基因全长蛋白的商业化抗体识别序列,也包含在AKT3-174aa中(图4B)。虽然全长蛋白和AKT3-174aa都可以被抗体识别,但由于两者长度差别巨大,在western blot的图中可以清晰地识别和区分出两种蛋白的对应条带(图3C)。
不要小看这个“巧合”,有现成可用的商业化抗体,可以大大节省你后续分子实验的时间成本和费用成本。因此,在选择1个环状RNA是否值得切入前,除了考虑它的生物学潜力(例如,是否来自明星基因)外,也需要多考虑它后续实验的潜在难度。调查候选环状RNA多肽是否有现成的商业化抗体,是你需要考虑的因素之一。
(2)竞争性抑制是最简单的机制研究切入点
环状RNA翻译的蛋白/多肽,往大了说,是一种新型蛋白,对发文章有新意加成的价值。往小了说,本质是一种来源RNA特殊可变剪切的截断蛋白。蛋白变异导致的功能变化,虽然会产生无数种可能,有很多方向值得探索。但对我们发文章来说,还是思路简单,套路可复制才是性价比最佳的选择。
如果环状RNA编码的多肽包含有母基因全长蛋白的核心关键序列,那么截断版的环状RNA多肽就会与母基因全长蛋白产生竞争性抑制关系(抢夺共同的互作蛋白)。例如,AKT3-174aa和母基因全长蛋白都含有308位置的可以与PDK1结合的可磷酸化位点,从而两者产生抢夺PDK1的效应。
这里有两个要点:
1)截断版的环状RNA多肽,由于序列短结构域单一,所以其在与母基因全长蛋白抢夺特定互作蛋白的时候,更高效。
2)截断版的环状RNA多肽没有母基因全长蛋白那样完整的序列,所以不具有母基因全长蛋白的完整功能。
所以环状RNA多肽颇有“占着茅坑不拉屎”的味道,抢了与母基因互作的蛋白,但自己又不能发挥母基因的完整功能,必然导致全长蛋白参与的通路发生变化。以这篇文章为例,AKT3-174aa抢夺了PDK1,导致全长蛋白AKT3无法与PDK1结合,那么ATK3无法完成磷酸化激活,进而导致AKT3的下游通路无法被激活。
这个套路思路清晰,证明起来也相对简单。当你没有特别好的思路的时候,这个方向是不错的切入点。
小结
在我们公众号的文章中,一直强调“干湿结合”。干实验,就是利用新组学和生物信息学,为研究打开局面,寻找创新性切入点。湿实验,就是用传统分子生物学手段稳扎稳打,将创新性的假设变成有说服力的实锤。这样子的思路,才能高效地实现高水平文章产出。翻译组、m6A RNA甲基化、单细胞等这些新兴的组学技术,是目前较为容易找到创新点的方向,值得你关注。
参考文献:
[1] Hansen T B, Jensen T I, Clausen B H, et al. Natural RNA circles function as efficient microRNA sponges[J]. Nature, 2013, 495(7441): 384.
[2] Memczak S, Jens M, Elefsinioti A, et al. Circular RNAs are a large class of animal RNAs with regulatory potency[J]. Nature, 2013, 495(7441): 333.
[3] Pamudurti N R, Bartok O, Jens M, et al. Translation of circRNAs[J]. Molecular cell, 2017, 66(1): 9-21. e7.
[4] Yang Y, Gao X, Zhang M, et al. Novel role of FBXW7 circular RNA in repressing glioma tumorigenesis[J]. JNCI: Journal of the National Cancer Institute, 2018, 110(3).
[5] Zhang M, Huang N, Yang X, et al. A novel protein encoded by the circular form of the SHPRH gene suppresses glioma tumorigenesis[J]. Oncogene, 2018, 37(13): 1805.
[6] Zhang M, Zhao K, Xu X, et al. A peptide encoded by circular form of LINC-PINT suppresses oncogenic transcriptional elongation in glioblastoma[J]. Nature communications, 2018, 9(1): 4475.




来第一个抢占沙发评论吧!