1.1 化学合成环状RNA
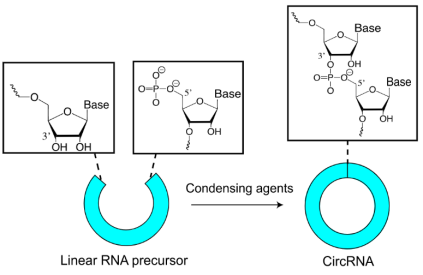
目前环状RNA的化学合成技术主要以磷酰胺(phosphoramidite)化学和固相合成为基础,并以天然的核苷三磷酸酯衍生物为原材料来进行制备的。这些衍生物采用非活性保护基团取代天然核苷三磷酸酯中的反应性氨基和羟基,能够实现3’-5’磷酸二酯连接并去除保护性基团,同时能最大程度避免2’-5’磷酸二酯键的形成及其它副反应的发生。后续经过合成后的处理和纯化,就可以生成高纯度的寡核苷酸。然而,目前的环状RNA化学合成技术只能合成少于70~80个核苷酸(nt)长度的环状RNA(图1)。
图1:体外化学合成环状RNA
1.2 酶促连接合成环状RNA
酶促连接合成通常通过体外转录(IVT)反应实现,该反应需要DNA模板、反应缓冲液和噬菌体RNA聚合酶。噬菌体RNA聚合酶通常来源于T7、SP6或T3噬菌体,其中较常见的是T7 RNA聚合酶。IVT反应允许以较低的成本进行更长的环状RNA合成,因此酶促合成是目前主流的环状RNA合成方法。但需注意的是,野生型噬菌体聚合酶自带的run-off性质可能导致合成不完整的RNA序列,因此有些研究通过突变野生型噬菌体RNA聚合酶来改善体外转录的质量,减少副反应的发生。
由于酶促连接是现阶段使用较多的体外合成环状RNA的方法,因此下文将对酶促合成进行重点介绍,而酶促连接又可进一步分为T4连接酶连接和核酶连接两种。
1.2.1 T4连接酶合成环状RNA
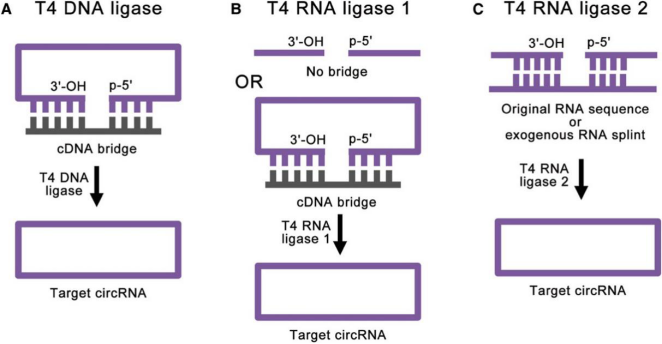
来源于噬菌体T4的多种连接酶都可以催化RNA连接反应,这其中包括T4 DNA连接酶(T4 Dnl)、T4 RNA连接酶1(T4 Rnl 1)和T4 RNA连接酶2(T4 Rnl 2)。当然,这种连接需要线性RNA前体上的受体区域(acceptor substrate)有3’-OH,且供体区域(donor substrate)有5’单磷酸才能进行。
1) T4 Dnl连接
T4 Dnl可以连接双链DNA或DNA/RNA杂交链(图2A),该方法通常需要利用一个与连接处互补的DNA(cDNA)模板或cDNA“桥”(bridge)来实现。这种方法的优点是连接的准确性较高,但是它要求线性RNA前体在连接处没有明显的RNA二级结构,且在双链区不能有高比例的尿嘧啶(U)。此外,T4 Dnl针对DNA/RNA杂交链的连接效率低于双链DNA(dsDNA)连接,因此实际上只有少数研究会使用该方法进行RNA连接。
2) T4 Rnl 1连接
T4 Rnl 1是一种比较常见的单链RNA(ssRNA)连接酶,它可催化单链RNA末端的3’-OH对活性的5’端进行亲核攻击,形成共价的3’-5’-磷酸二酯键,进而形成环状RNA(图2B)。有些研究也会在此过程中使用cDNA“桥”来防止线性RNA前体折叠成不必要的二级结构。值得注意的是,T4 Rnl 1对5’末端和3’末端的核苷酸明显偏好性:其对3’末端的核苷酸偏好为A>G≥C>U,而对5’末端核苷酸偏好则为pC>pU>pA>pG。
该连接方法的优点是仅可以合成极短的环状RNA(短至6~8 nt),但对于较大的RNA分子的连接效率会显著降低。与T4 Dnl类似,线性RNA前体末端若具有明显的RNA二级结构会大大降低T4 Rnl 1的连接效率。此外,该反应还存在分子间末端连接(低聚物)的严重副反应,该副反应会随着线性RNA前体浓度的增加而增加,并会限制环状RNA的产量。
3) T4 Rnl 2连接
T4 Rnl 2与T4 Rnl 1相似,它也可催化RNA末端的3’-OH与5’末端形成共价的3’-5’-磷酸二酯键。不过T4 Rnl 2在连接双链RNA(dsRNA)时要比连接单链RNA厉害得多(图2C)。基于这一特点,当线性RNA前体折叠成二级结构,即接头区域为双链结构时,T4 Rnl 2的连接效率要比T4 Rnl 1高得多;而且利用外源RNA“夹板”序列(RNA splint),T4 Rnl 2还能实现对单链RNA末端的连接。
不过此方法不能用于连接较短的RNA前体,这是因为当线性RNA前体的长度短于30 nt时,其连接处形成的复合物是不稳定的。与T4 Rnl 1一样,T4 Rnl 2也存在对大片段RNA分子连接效率低和副反应多的问题。
综上,T4 Dnl和T4 Rnl 1适用于没有复杂二级结构的RNA环化;而T4 Rnl 2更适用于接头区域为双链的线性RNA前体,因此在实际操作中需要根据线性RNA前体的二级结构来选择不同的T4连接酶。需注意的是,所有这些连接酶合成方法都不能实现大片段RNA分子的环化,也不能完全避免分子间末端连接的副反应。
图2:T4连接酶合成环状RNA的策略
1.2.2 核酶合成环状RNA
1) I型内含子自剪接系统(Group I intron self-splicing system)
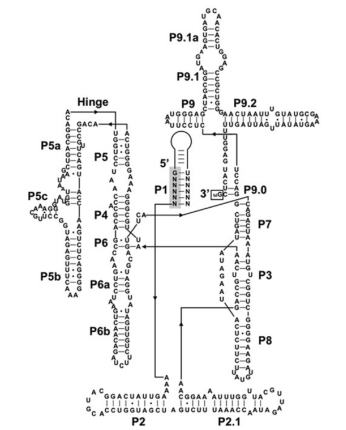
1982年,科学家在四膜虫(Tetrahymena thermophila)的rRNA转录本中首次发现了I型内含子,它同时也是第一个被发现的核酶——无需任何蛋白质的帮助就可以帮助初级转录本RNA进行自我剪接(self-splicing)。自那以后,在许多生物和病毒基因组的tRNA、mRNA和rRNA中也陆续发现了很多能够自我剪接的I型内含子。尽管这些I型内含子来源于完全不同的初级转录本,但它们的催化中心结构却惊人得相似。根据二级结构可将I型内含子划分为10个结构域,并分别命名为P1~P10,其催化中心位于P4-P6(P4, P5, P6)和P3-P9(P3, P7, P8, P9)结构域的交界处,P1和P10则分别为5’和3’剪接位点(splice site),而其他围绕着保守的催化中心的序列则负责维持核酶的稳定性和正确折叠(图3)。
图3:Tetrahymena thermophila I型内含子的二级结构示意图
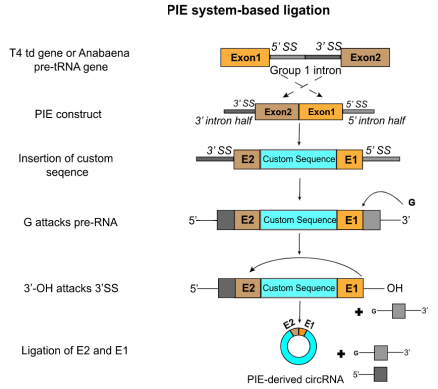
利用I型内含子自剪接的特点进行改造的体外合成环状RNA系统正是目前最为常见的体外合成环状RNA的技术,该技术也被称为PIE法(permuted introns and exons),只需GTP和Mg2+就能实现RNA的连接(图4)。
图4:PIE法制备环状RNA的原理
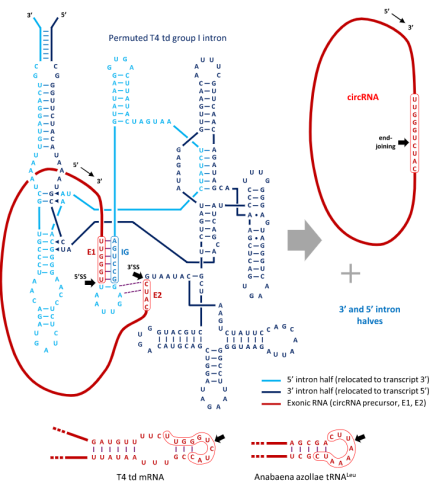
用于PIE合成的体外转录模板通常采用来自Anabaena tRNALeu基因或T4噬菌体的胸苷酸合成酶(thymidylate synthase, td)基因上的I型内含子序列,这两种I型内含子都已被证实可以高效促进RNA的环化。
以td基因内含子改造的PIE系统为例(图5),它主要包含td内含子的5’端内含子序列(5H)、3’端内含子序列(3H)、相邻外显子序列(E1和E2)、内部引导序列(internal guide sequence, IG)、待环化的RNA(RC)以及为促进折叠和稳定内含子而添加的末端同源臂序列。
RNA的环化过程大致如下:
(1)一个鸟苷亲核物插入RC-5H交界处(5’ SS)并置换RC的3’末端;
(2)该鸟苷继续进一步插入3H-RC交界处(3’ SS),在与RC 5’末端连接时取代3H的3’末端,从而形成环状RNA。
由于该催化反应的关键是E1和E2与内含子序列发生碱基互补配对,因此E1和E2序列也都会被纳入新生的环状RNA中。
图5:I型内含子PIE系统的二级结构示意图
与化学连接和酶促连接相比,PIE方法可用于较大片段的线性RNA前体环化,而且其反应条件和纯化方法也比较简单。现有的研究已经可以通过设计特定的PIE转录模板对几乎任何序列的线性RNA前体进行高效的环状RNA合成。基于以上优势,PIE法是目前研究最多和使用最广泛的RNA连接方法。
然而PIE方法仍存在明显缺陷:
1. 由于RNA二级结构的复杂性,不同的RNA序列会导致最终形成的环状RNA结构存在巨大差异;
2. 此外,由于外源外显子序列的引入,最终形成的环状RNA序列将与原始的线性RNA前体序列不同,这可能会对环状RNA的功能验证产生负面的影响。
2) II型内含子自剪接系统(Group II intron self-splicing system):
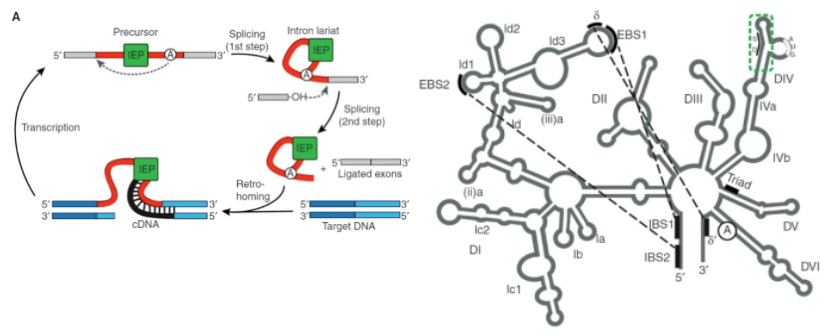
II型内含子(Group II introns)是一类发现于细菌和细胞器基因组中的可移动遗传物质,它们被认为是真核生物中剪接体内含子和逆转录转座子的祖先,其包含一个具有催化活性的内含子RNA(“核酶”)和一个内含子编码蛋白(intron-encoded protein,IEP),两者共同介导内含子在基因组内进行增殖。II型内含子RNA通过与剪接体内含子类似的转酯反应催化其自身的剪接,产生拼接的外显子和一个切下来的内含子套索RNA(lariat RNA)。IEP是一种多功能的逆转录酶(reverse transcriptase, RT),它能通过稳定具有催化活性的RNA结构来协助剪接。在剪接后,IEP还会与切除的内含子RNA相连,组成一种可以入侵基因组DNA的核糖核蛋白(Ribonucleoprotein, RNP)(图6A)。
II型内含子RNA同样具有保守的二级结构,其长度为400~800 nt,大致可分为六个区域DI-DVI(也可称为D1-D6),它们从结构上来看就像一个“车轮”(图6B)。其中DV是活性位点的核心,包含所谓的催化三碱基(catalytic triad)“AGC”和一个“AY”(Y代表碱基C或T)凸起,这两者都能结合具有重要催化作用的Mg2+离子;而DVI包含一个隆起的腺苷作为分支位点(branching site),其2’-OH基团用于亲核攻击内含子5’剪接位点上的磷酸基团,起始剪接反应并形成套索RNA。DI是最大的结构域,包含上下两半部分,其中的EBS1可以与5’剪接位点交界处的内含子结合位点(IBS1)配对,以确定剪接的确切位置;DII和III是两个较小的结构域,对组装活性内含子结构和激活剪接反应方面起着关键作用,其中DIII还可作为“催化效应器”来加速催化反应;DIV是一个茎环(stem-loop)结构,其loop序列上包含maturase(成熟酶)的ORF,靠近其5’端的亚结构域DIVa含有一个对IEP具有高亲和力结合位点(图6B)。II型内含子RNA还具有保守的5’和3’端序列,分别是GUGYG和AY,其与剪接体内含子的保守序列相似(即GU. . . AG)。
图6:II型内含子的工作原理及二级结构示意图
II型内含子序列上的一系列保守结构域(包括由希腊字母命名的结构域、外显子结合位点EBS和内含子结合位点IBS)还能通过远距离的三级相互作用形成保守的三级结构,进而将相距甚远的序列凝聚在一起形成新的活性部位,该活性部位会结合剪接位点和分支点(branch-point)的核苷酸残基,然后在Mg2+的帮助下来催化连接反应。
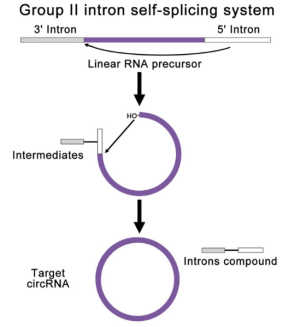
科学家们基于II型内含子的自催化特性开发出了另一种体外合成环状RNA的PIE技术——利用反向剪接反应(inverse splicing)将一段RNA末端的5’剪接位点与该RNA的3’剪接位点连接起来形成环状RNA(图7)。
图7:II型内含子PIE系统的原理图
中科院上海营养与健康研究所的王泽峰研究员课题组及其创立的环码生物医药公司在基于II型内含子环化特性开发的体外环化技术以及环状RNA的体外翻译机制领域就取得了较大突破。
他们于2022年发表了一个基于II型内含子环化且适用于大规模生产的体外RNA环化新技术,该技术可以实现高效共转录环化RNA的同时又能避免掺入无关序列,而且该环状RNA可以在细胞和小鼠体内进行高效翻译的同时又不会引起严重的先天性免疫反应。
具体来说,他们是对来自破伤风杆菌表层蛋白(surface layer protein of Clostridium tetani, ctSLP)的一个II型内含子上的某个loop区域进行了切割,构建了一种分裂的内含子(split-intron)环化系统,然后将需要环化的序列插入到两段内含子中间,该环化序列两端还分别包含两个6 nt的短序列IBS3和IBS1。
在体外转录时,RNA前体会通过II型内含子介导的自我剪接反应产生一个包含目的序列的环状RNA和一个分支状内含子RNA(branched intron RNA),该系统也被他们命名为“CirCode”,可以作为一个通用的平台来生产任何指定序列的环状RNA并用于蛋白质翻译(图8)。
图8:环码生物体外环化技术CirCode系统的工作原理示意图
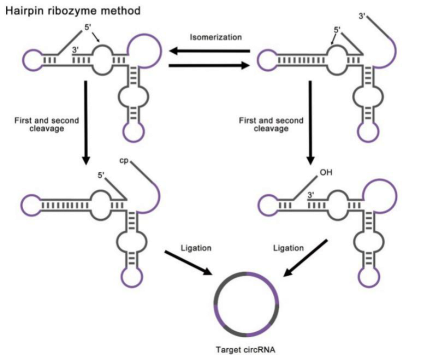
3) 发夹状核酶(Hairpin ribozyme, HPR)
HPR可以单链环状DNA为模板通过滚环(rolling circle)反应和自剪接反应产生环状RNA。带有HPR的线性RNA前体会折叠成两种具有切割活性的构象以暴露出3’端和5’端,进而通过自发连接成环状RNA(图9)。
这种方法可以高效生产较短的环状RNA,但它的劣势在于由HPR催化的裂解和连接是一种动态平衡,这会导致形成的环状RNA并不稳定;此外,生成的环状RNA中会包含HPR序列,可能对环状RNA的功能产生不利影响。
图9:核酶合成环状RNA的策略
综合来看,在以上所有酶促连接合成环状RNA的方法中,PIE法显示出较明显的优势,并且被广泛用于基础研究和工业生产中(表1)。
但值得注意的是,不同环化策略合成的环状RNA所触发的人体细胞免疫反应程度存在较大差异:中国科学院分子细胞科学卓越创新中心的陈玲玲团队于2021年报道,由I型内含子自剪接系统合成的环状RNA会分别引入~74 nt(td)或~186 nt(Anabaena)的外源序列,这些外源序列会触发较强的先天性免疫反应;相比之下,由T4 RNA连接酶合成的无外源序列掺入的环状RNA表现出最小的免疫原性。
表1体外合成环状RNA的方法对比
参考文献
[1] Chen R, Wang S K, Belk J A, et al. Engineering circular RNA for enhanced protein production[J]. Nature Biotechnology, 2022.
[2] Liu C-X, Chen L-L. Circular RNAs: Characterization, cellular roles, and applications[J]. Cell, 2022.
[3] Chen X, Lu Y. Circular RNA: Biosynthesis in vitro[J]. Frontiers in Bioengineering and Biotechnology, 2021.
[4] Liu X, Zhang Y, Zhou S, et al. Circular RNA: An emerging frontier in RNA therapeutic targets, RNA therapeutics, and mRNA vaccines[J]. Journal of Controlled Release, 2022.
[5] Lee C H, Han S R, Lee S-W. Group I Intron-Based Therapeutics Through Trans-Splicing Reaction[J]. Progress in Molecular Biology and Translational Science, 2018.
[6] Belfort M, Lambowitz A M. Group II Intron RNPs and Reverse Transcriptases: From Retroelements to Research Tools[J]. Cold Spring Harbor Perspectives in Biology, 2019.
[7] Lambowitz A M, Zimmerly S. Group II Introns: Mobile Ribozymes that Invade DNA[J]. Cold Spring Harbor Perspectives in Biology, 2011.
[8] Chen C, Wei H, Zhang K, et al. A flexible, efficient, and scalable platform to produce circular RNAs as new therapeutics[J]. bioRxiv, 2022: 2022.05.31.494115.
[9] Obi P, Chen Y G. The design and synthesis of circular RNAs[J]. Methods, 2021, 196: 85–103.





来第一个抢占沙发评论吧!