与DNA甲基化一样,科研工作者对RNA甲基化研究也经历了相当长的时间。高通量技术的普及推动了表观转录组学(Epitranscriptomes)的发展,并使其成为新的研究热点。RNA甲基化主要包括N6-methyladenosine(m6A),5-methylcytidine(m5C)和ribose methylations。
m6A修饰参与调控了RNA许多重要生物学功能,包括肥胖、神经突触信号传递、肿瘤、生殖发育等;在分子层面,m6A修饰主要参与调控RNA-蛋白相互作用,RNA二级结构稳定性,RNA剪接,翻译调控等。在细胞内,m6A修饰主要由YTH domain家族蛋白识别,包括YTHDF1,YTHDF12和YTHDF13。
除了线性mRNA存在甲基化修饰外,最近研究表明环状RNA也同样受到甲基化的修饰,并具有一定的特点[1,2]。本周解读一篇文章,研究环状RNA甲基化以及对蛋白翻译的调控,我们可以借鉴其中的高通量测序和生物信息学方法。
研究背景
本文作者在2015年报道含有Internal ribosomal entry site(IRES)的circRNAs能够在真核细胞内编码蛋白(戳这里查看~)。接着偶然发现不含IRES的阴性对照组circRNAs也能在人类细胞中翻译,预示着可能还有其他机制调控着环状RNA翻译。
研究思路和结果
作者利用多种方法研究环状RNA甲基化和翻译机制,主要包括:
1. 发现候选circRNAs的共有序列特征,推测甲基化驱动翻译
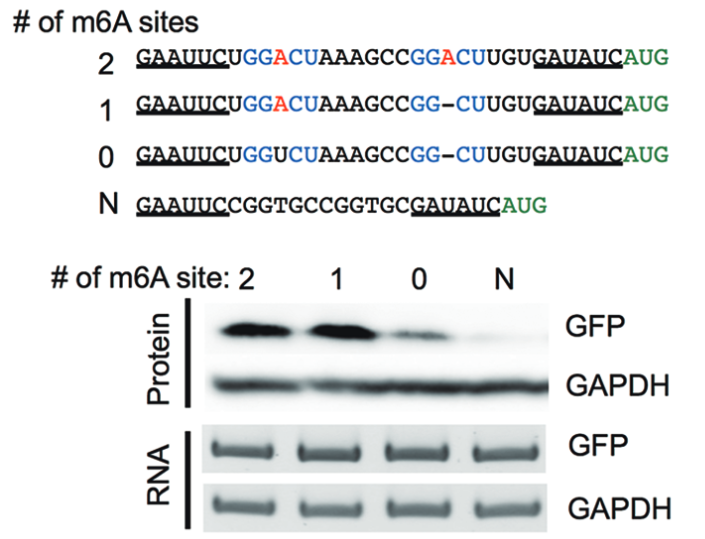
作者对不含IRES的circRNAs的翻译起始位点附近序列进行序列比对,在起始密码子附近都发现有RRACH片段(R= G or A; H = A, C or U),能够被m6A修饰,简称(RRACH motif)。作者推测RRACH片段的m6A修饰驱动了circRNAs的5’帽子非依赖性的翻译(图1)。
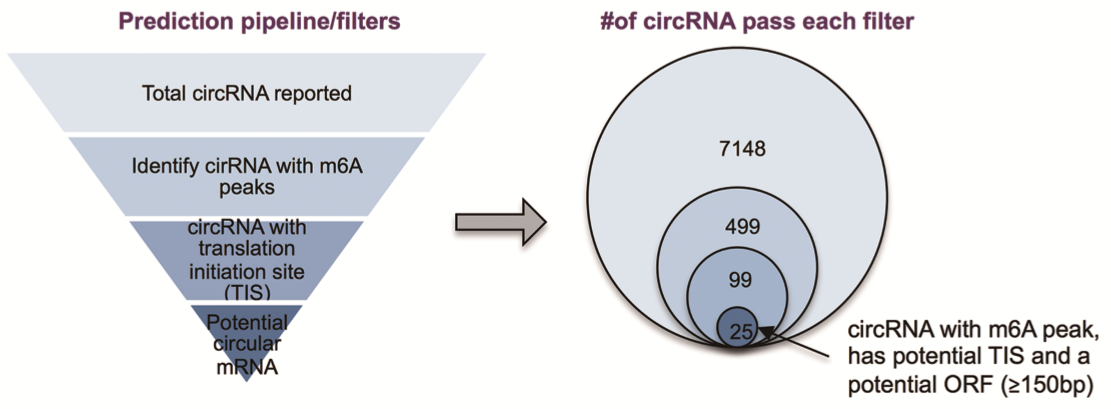
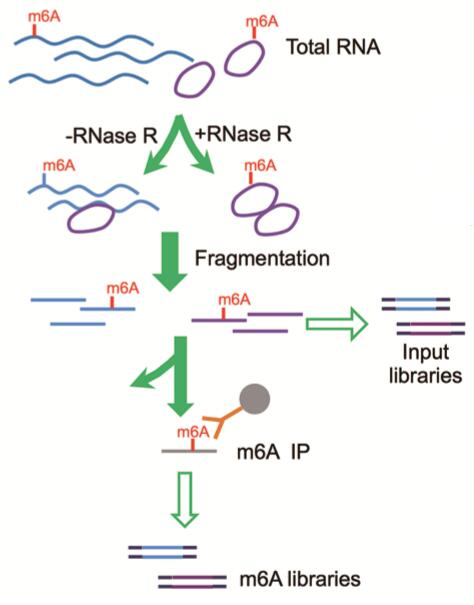
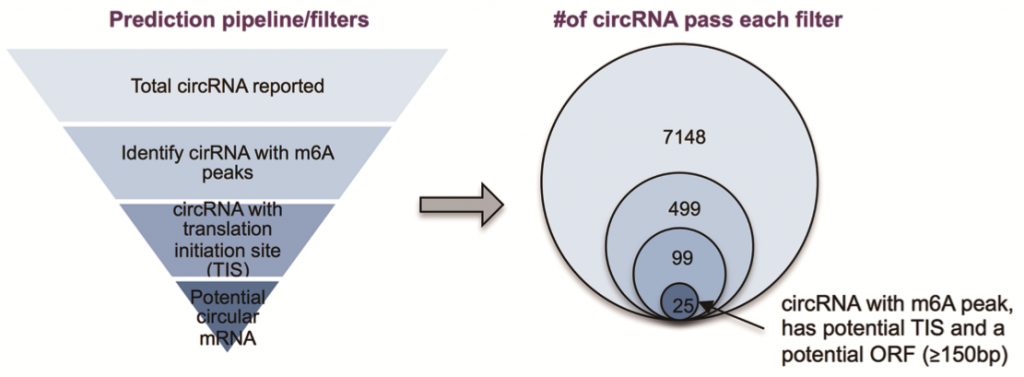
基于上述推论,作者通过甲基化环状测序circRNA-m6A-seq(m6A-IP富集的环状测序),筛选甲基化修饰的内源性circRNAs,再利用生物信息学筛选含有长ORF(≥150 nt)具有翻译潜力的circRNAs。
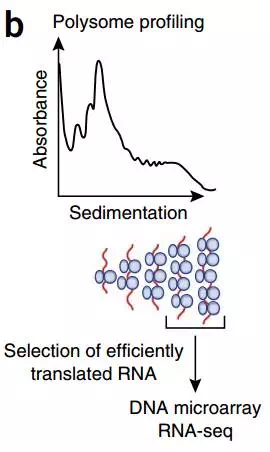
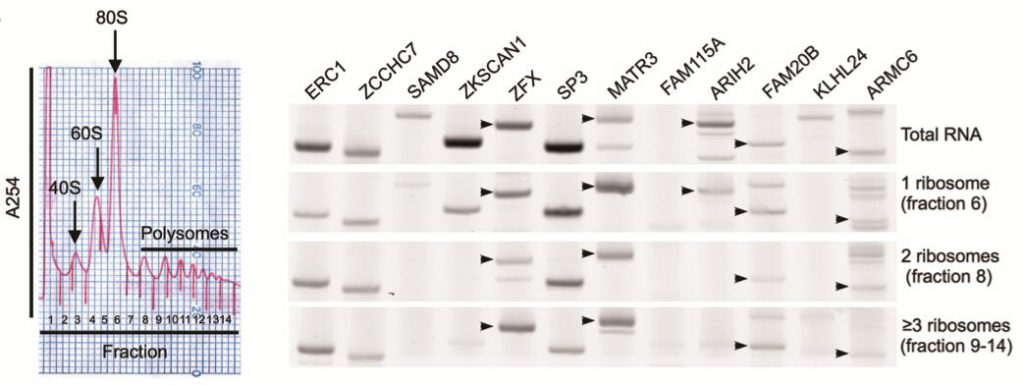
3. Polysome表达谱验证候选circRNAs与核糖体相关
Polysome表达谱根据结合核糖体数目,将多核糖体组进行分离,然后研究与其结合的RNA。通过翻译组学的技术,结合circRNA生信流程,检测上述筛选的25个circRNAs是否与Polysome结合。结果显示有10个circRNA与Polysome结合,显示出翻译的潜力。
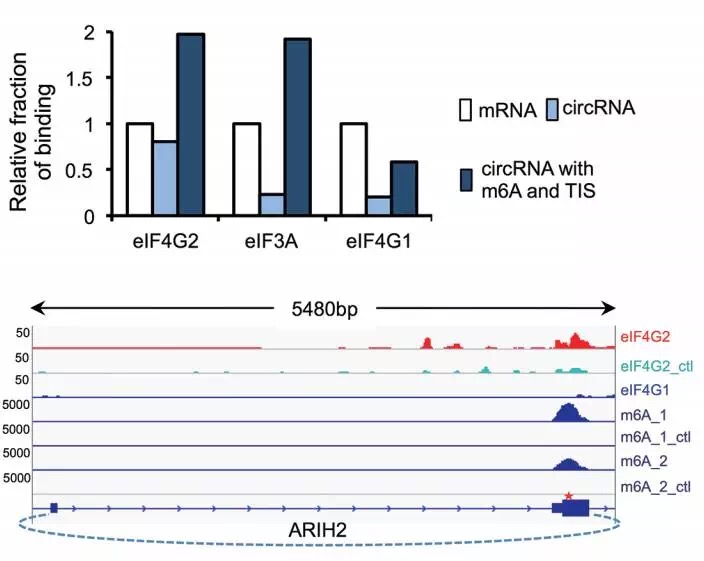
通过对数据库中人源细胞CLIP-seq dataset(ENCODE)、m6A profiles [3]、翻译起始位点图谱(TIS)[4]进行分析,计算线性和环状RNA与甲基化相关基因(eIF4G2,eIF3A /eIF4G1)的结合频率等参数。结果发现,线性与elF家族结合频率较高。然而,含有m6A和TIS环状RNA与elF家族结合频率比线性RNA高。说明eIF家族偏好性结合环状RNA的m6A和TIS位点,可能驱动环状RNA的eIF4G2依赖性翻译。
来源:基迪奥生物





来第一个抢占沙发评论吧!