circlncRNAnet: An integrated web-based resource for mapping functional networks of long or circular forms of non-coding RNAs
Gigascience 2017.11.29
app:http://app.cgu.edu.tw/circlnc/
一、简介
lncRNA和circRNA 虽然并不编码蛋白,但是其在基因调控中起到重要的作用。
这些非编码RNA通常通过表观修饰和转录后调控行驶功能。
考虑到他们靶基因的广泛性,因此深入的了解他们必须建立在更为整合的数据基础上。
今天给大家推荐一个研究神器circlncRNAnet,可以实现对ncRNA的一站式操作,文章今年11月29日发表在Gigascience上,作者单位是台湾长庚纪念医院。
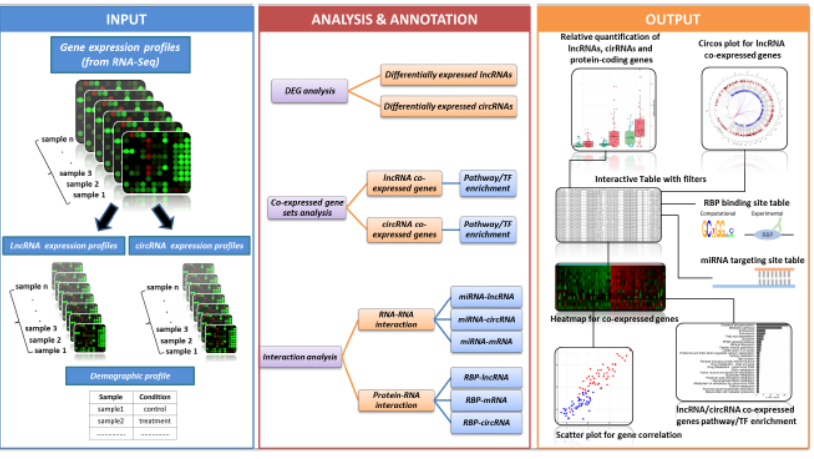
二、数据和功能
1、数据输入
数据上传部分有两个单独的页面(lncRNA、circRNA),用户可以上传用featureCounts生成的以tab分割的RNA原始read count文件。另外指定样本的条件文件。
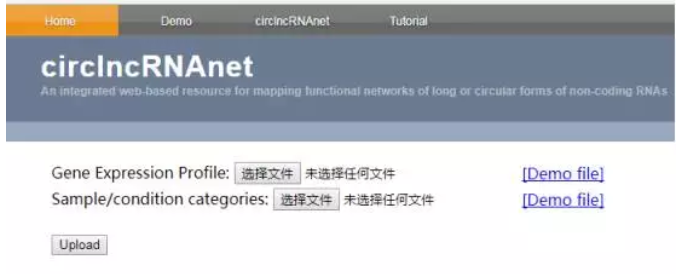
lncRNA上传文件页面示例

circRNA上传示例
当你成功的上传一个文件之后,后台会自动进行格式转化,差异表达分析,蛋白互作网络分析,并且自动生成报告,并显示进度条。
2、分析模块介绍
Analytic module #1:coding–non-coding co-expression (CNC) network profiling
数据上传之后会首先调用DESeq2进行差异表达分析,这里需要用户指定fold change值和p-value值。
然后利用WGCNA包进行计算差异的circRNA或者lncRNA与用户上传的所有的基因的皮尔逊相关系数。相关性较高的基因将会进行通路富集分析。
Analytic module #2: RBP interactome mapping
由于lncRNAs经常会与一些RNA绑定蛋白或者染色体修饰蛋白相关,因此为了深入的研究这一部分。
我们利用MEME预测RBP binding motifs,然后从GENCODE中得到所有的lncRNA的序列,然后利用FIMO进行寻找RBP绑定位点,同时我们还整合了PIP-seq数据。
Analytic module #3: ceRNA networking
在线做ceRNA网络,想想都激动。
为了实现这个功能,我们在服务器上安装了一个基于序列的分析lncRNA miRNA海绵结构的工具。考虑到miRNA3端匹配不好的问题,我们还利用三种不同的miRNA靶基因预测工具来进行预测。(RNAhybrid, miRanda and TarPmiR)
Analytic module #4: Multi-tier regulatory hierarchy
分析模块4可以实现ncRNA-miRNA-mRNAs 分级调控,会生成一个二级网络。
三、开发语言和项目概况
Project name: circlncRNAnet
Project home page: http://app.cgu.edu.tw/circlnc/ [72],
https://github.com/smw1414/circlncRNAnet [73]
Operating system(s): Platform independent
Programming language: PHP, JavaScript, R, R shiny and Shell script
Other requirements: JavaScript supporting web browser
License: GPLv3
Research Resource Identifier: circlncRNAnet, RRID:SCR_015794
三、网站可以做的功能总览

四、示例展示
除了上传自己的数据之外,网站还与TCGA数据库实现共享(不知道是不是实时的)
目前有20中癌型,大家可以上去点一点试一试。

看看有没有样本信息,如果没有就是用的TCGA中所有的信息喽。(这里我没找到线索,或者有,我没看到。)
选择某个癌型,然后经过筛选P和FC进行筛选差异
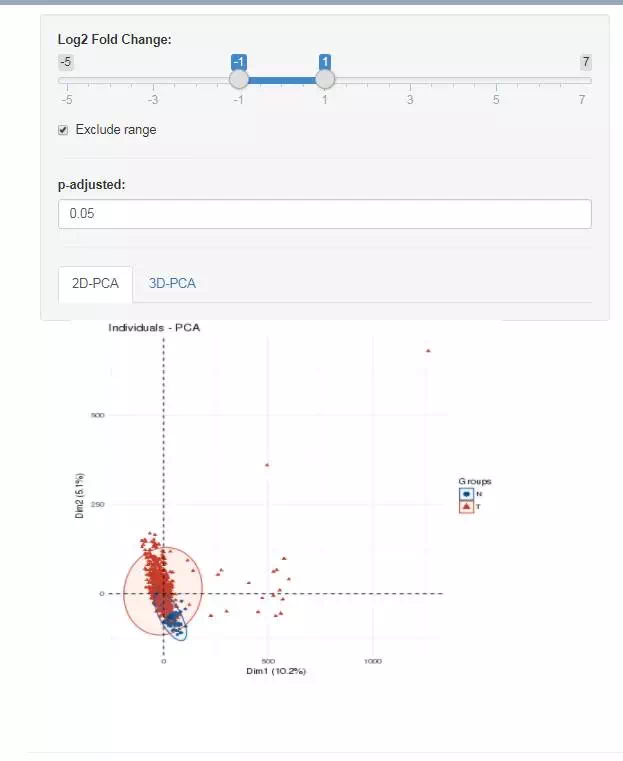
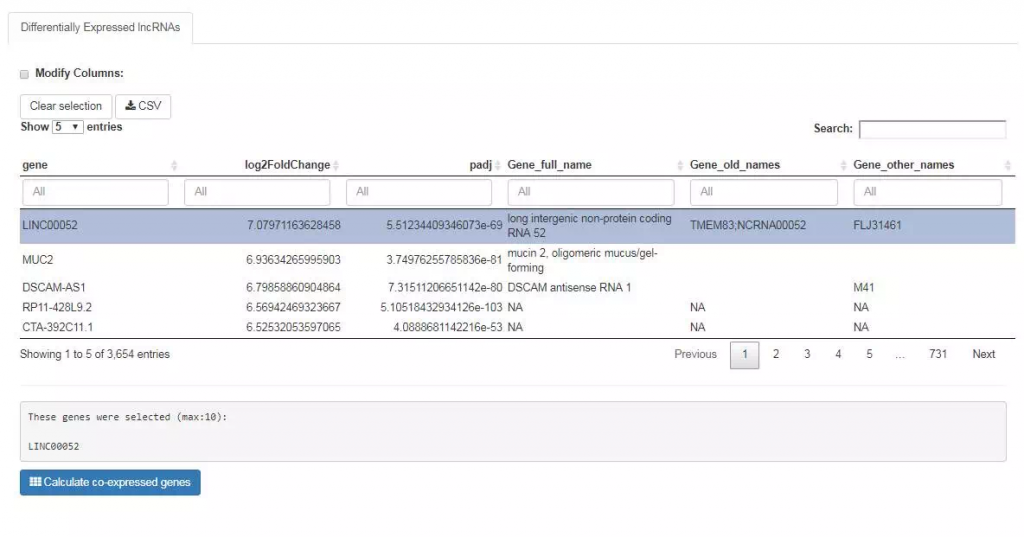
点击,自动进行共表达网络构建
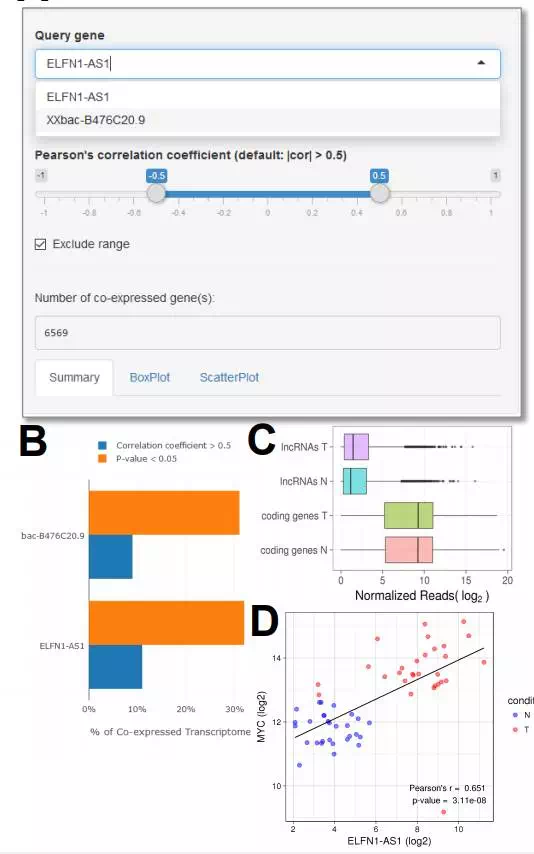
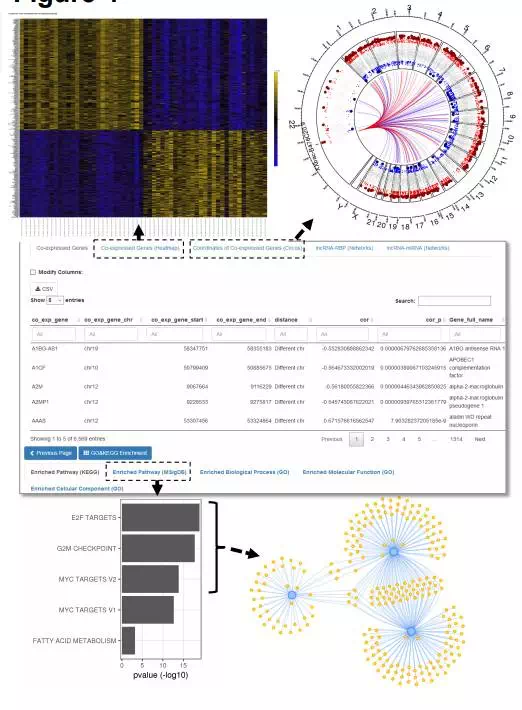
更多牛叉的功能,等着你自己去发现,当然这一个网站可能不能实现你所有的分析
文章:生信人(微信公众号)





来第一个抢占沙发评论吧!