CircRNA引物设计
与常规PCR引物设计不同,circRNA引物设计应满足以下原则:
1、对于外显子环化circRNA,引物跨剪切位点(backsplice)设计;
2、对于内含子环化circRNA,可跨剪切位点设计,也可围绕内含子区域设计引物;
3、扩增产物长度建议不超过100 bp。
一、序列位置变换
由于已获取的序列为环序列从剪接位点(backsplice)处打开后的线性序列,因此,为复原序列成环时的位置关系,应先进行序列位置变换后再进行引物设计(设计原理如下图)。
二、截取3’端50-100 bp长度序列置于5’端头部,形成一个新的序列。以hsa_circ_0063162为例,具体步骤如下:
hg19_hub_1_salzman_circRNAsrange=chr22:36889680-36889850
5′ CTGCATAGAAGCTTCGCAAATCGGTGTCCAAACAGTTCTCCAGGAAGCGCCTCAGAGGTAGGATGTGCCCGATCGGGGCAGTCCAGTCTGTTAAGAAGTCTTCCACGATGTGATGGATGGCCGTGGGCCGAGGCAGGGGCCAGTGTGCAGGGCGGAACCTCACCTCCTGTT 3’
三、将3’端蓝色序列移至序列5’端头部,位置变换后的新序列如下:
5′ TTAAGAAGTCTTCCACGATGTGATGGATGGCCGTGGGCCGAGGCAGGGGCCAGTGTGCAGGGCGGAACCTCACCTCCTGTTCTGCATAGAAGCTTCGCAAATCGGTGTCCAAACAGTTCTCCAGGAAGCGCCTCAGAGGTAGGATGTGCCCGATCGGGGCAGTCCAGTCTG 3′
四、新的序列中蓝色字体与黑色字体碱基相连处即为circRNA剪接位点处,引物设计按照常规PCR引物设计原理,跨剪接位点上下游进行设计,最终扩增产物必须包含剪接位点。
五、Primer 5引物设计步骤
打开 primer 5.0 软件,点击“File”/New/DNA sequence,如下图所示:
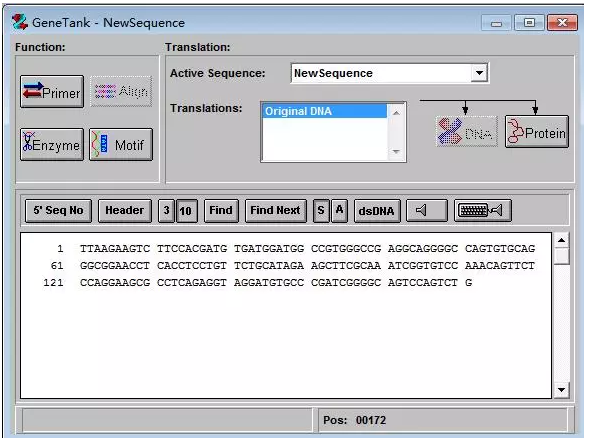
六、复制粘贴新的circRNA序列至软件中,如下图:
七、点击 primer按钮进行引物设计,如下图:
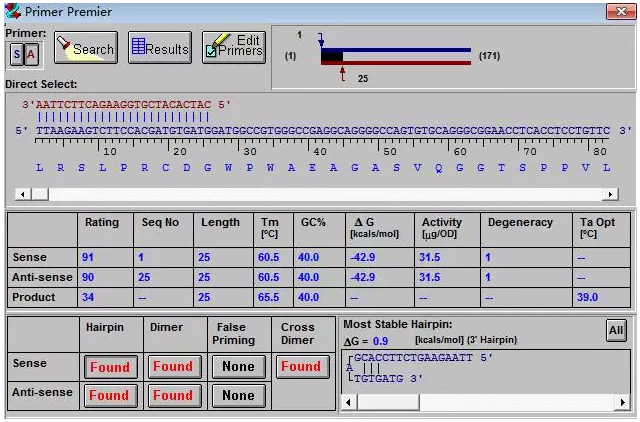
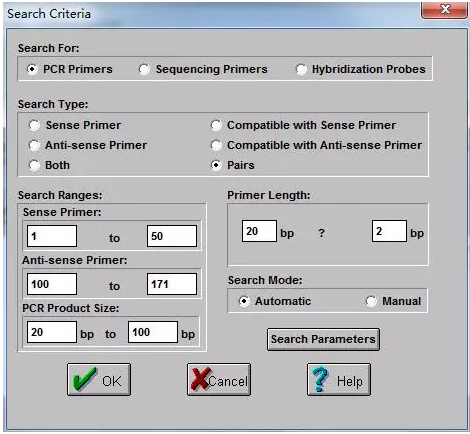
八、点击 search按钮,程序会自动设计引物,系统弹出下图对话框。请在以下对话框中修改参数,包括正反向引物结合模版起始终止位点以及PCR产物。
以hsa_circ_0063162为例,剪接位点处于77-86之间,sense primer:1-50;Anti-senseprimer:100-171;PCR Product:20-100 bp。
九、引物筛选
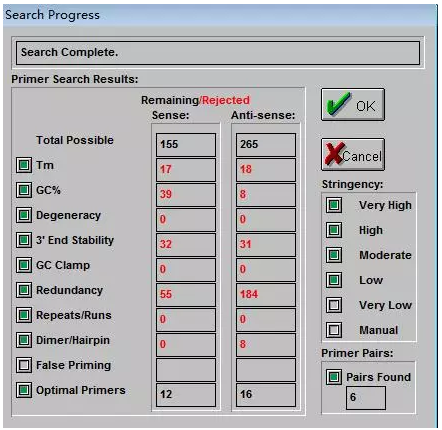
填写以上参数后,点击OK按钮,程序会弹出引物Search结果,如下图所示,共有6对引物符合要求。
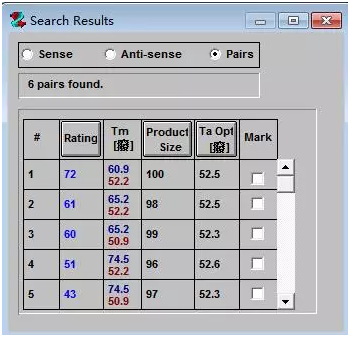
点击OK按钮,程序会弹出可选引物对的评分,如下图所示:
十、引物评估
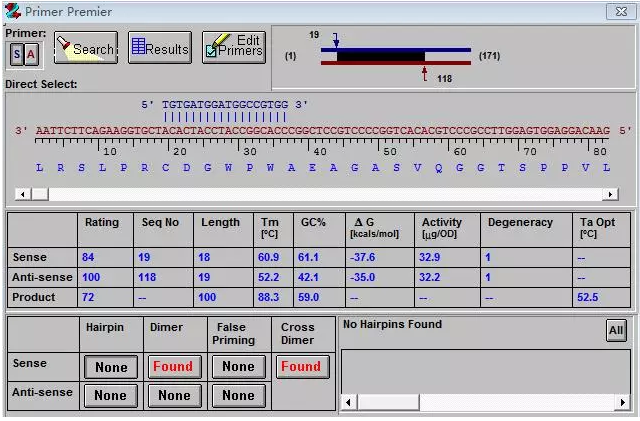
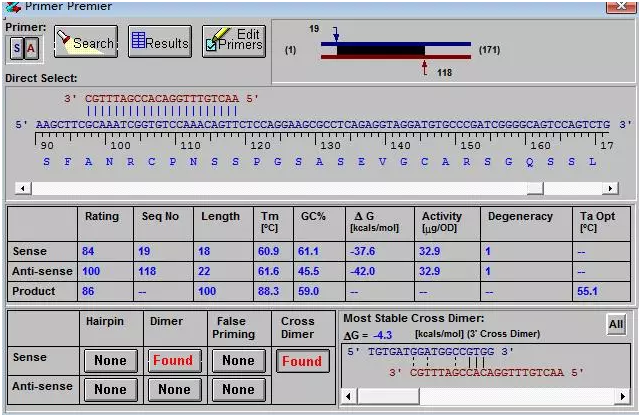
对软件给出的引物根据系统评分从高到低进行筛选。以hsa_circ_0063162为例,选取评分最高的引物进行评估,如下图所示:
十一、引物调整
使“Sense”中的“Tm”与“Anti-Sense”中的“Tm”差值的绝对值不大于2.5,“GC%”=40%~70%;核对“Hairpin”,“Dimer”,“False Priming”,“Cross Dimer”,调整后结果如下图所示:
16.引物输出
点击“Edit”/“Copy”,输出“Sense primer”及“Anti-sense primer”,引物序列信息如下:
Senseprimer :5′ TGTGATGGATGGCCGTGG 3’;
Anti-senseprimer:5′ AACTGTTTGGACACCGATTTGC 3′
进一步地,可再对获取的引物进行特异性评估,将引物序列导入NCBI数据库(http://blast.ncbi.nlm.nih.gov/Blast.cgi)中,采用“Primer-Blast”工具进行引物特异性比对分析。





选择spliced length,这才是circRNA的序列
在cirbase上面查到的话,会有spliced length,但是话说这个是怎么查看剪切后的序列的?整个序列是在position中点开查看的对吧
fasta
[/给力][/给力]
请问环状rna的blast有什么特殊的么?为什么我blast出来的不太对劲儿
不对劲是什么?