经典的分子生物学中心法则认为, 遗传信息(基因)通过转录从DNA传递到RNA, 再通过翻译从RNA传递到蛋白质; 在此过程中, RNA是遗传信息从DNA传递到蛋白质的中间体[1]。然而, 从上个世纪70~80年代起的研究发现, 遗传信息的传递在RNA水平也存在着广泛而又复杂的调控作用[2-3]。生命起源的RNA世界假说[4]和全转录组RNA表达分析结果[5]都显示基因表达在RNA水平存在着更复杂而又精细的调控作用(RNA complexity), 而且这种调控作用具有显著的时间和空间特性, 揭示RNA水平的调控可能是生命功能复杂性的核心所在[6]。
真核生物基因序列的不连续性和RNA剪接
真核生物蛋白编码基因在转录生成前体RNA后, 通常会通过RNA剪接将含有蛋白编码信息的外显子序列顺序地连接在一起, 形成成熟的线形mRNA分子, 而去除不需要的内含子序列。在高等真核生物(从果蝇到人类)中, 通过可变剪接反应可以由一个基因产生多种不同功能的成熟mRNA及其相应蛋白产物, 这在不改变基因数目的前提下极大地提高了基因表达及其功能的复杂性和多样性。RNA可变剪接与生命体正常的生理活动息息相关, 例如神经系统中的可变剪接极大地丰富了神经反应的多样性[7]。最新的研究结果表明, 至少90%的人类基因通过可变剪接由一个基因产生多种不同功能的成熟mRNA及其相应蛋白产物[5,8-9]。然而, RNA的异常可变剪接则导致多种重要人类疾病[10], 包括一些严重的遗传性疾病, 如由于SMN异常剪接导致的脊髓肌肉萎缩症(spinal muscular atrophy)、囊肿性纤维化(cystic fibrosis, 一种遗传性胰腺病)和强直性肌营养不良症(myotonic dystrophy)。因此, 对RNA可变剪接的正常生理功能和异常病理作用及其机制的研究一直是领域的热点。
反向剪接反应和环形RNA的产生
一般来讲, 正常的RNA剪接反应可以被简单地分为两步转酯反应: (1)位于分支位点的核苷酸(一般为腺嘌呤核苷酸, A)通过其2′-羟基(2′-hydroxyl)攻击上游5′剪接位点, 将上游外显子从内含子上解离, 并形成含有套索结构(lariat)的内含子和下游外显子中间体; (2)位于上游外显子末端剪接位点的3′-羟基攻击下游剪接位点, 将上下游外显子顺序地连接在一起, 最终形成成熟的(线形)RNA分子, 而剩余的内含子套索结构RNA片段则被释放并快速降解[11]。然而, 在真核生物体内还存在特殊的反向剪接反应(back splicing), 使得下游外显子序列反向与上游外显子连接环化, 我们将这种反应称为反向剪接导致的外显子环化, 或者简称为“外显子环化(exon circularization)”, 其最终产物为环形RNA(circular RNA)。尽管在上个世纪就已经在哺乳动物细胞中[12-14]发现了外显子环化导致的环形RNA产生, 但是因为这一类环形RNA的表达量非常低, 并且被认为是一类由于错误剪接造成的剪接副产物[15], 推测它们可能没有任何生物学功能, 这样在随后的二十年里再也没有发现新的环形RNA。
新技术和新思路带来的环形RNA大发现
人类和其他模式生物基因组计划[16-20]的完成让我们认识到人类基因组所蕴含的蛋白编码基因不是原来推测的10万个, 而是只有2~3万个, 和果蝇(1.4万)、线虫(2万)等的编码基因相差无几。显而易见的是, 人类要比果蝇和线虫复杂的多, 这揭示物种间的差异不是简单的体现在基因数目差异的水平上, 而更多的是在基因表达调控的层面上。利用经典的、针对3′-末端含有poly(A)尾巴的mRNA分离纯化并结合高通量测序和计算分析, 科学家们获得了来自不同物种/组织等的详尽mRNA表达谱, 并进一步揭示了基因表达在可变剪接[5,8-9]和非编码调控[21-22]等水平的RNA复杂性调控。
然而, 由于技术和认识上的局限性, 一直以来对那些3′-末端不具有poly(A)尾巴的RNA都缺乏系统性的研究。直到2011年, 我们的研究团队发展了一种新的纯化方法, 可以有针对性地获得3′-末端不具有poly(A)尾巴的RNA, 并开展高通量测序和计算分析[23]。出乎意料的是, 我们在人源细胞系中发现了一些分别来自于内含子(excised intron)或外显子(excised exon)、3′-末端不具有poly(A)尾巴的RNA表达信号。进而, 我们在2012年和2013年分别报导了内含子来源的特殊结构长非编码RNA——sno-lncRNA[24]和内含子环形RNA[25]的存在。利用我们的数据[23], 其他课题组也报导了外显子环形RNA在人胚胎干细胞中的大量存在[26]。随后, 不同的课题组又在多个人/鼠细胞系中证实了外显子环形RNA的广泛存在[27-29], 并在生物信息学水平揭示了一些环形RNA存在的结构/序列基础。虽然这些突破性研究结果极大地推动了对环形RNA的认识, 但是外显子环形RNA产生的具体机制不详, 更为重要的是缺乏直接的实验证据。
互补序列介导的外显子环化和可变环化
为了深入研究外显子环化产生环形RNA的具体分子机制, 我们通过特殊核酸外切酶(RNase R)在3′-末端不具有poly(A)尾巴的RNA组分中对环形RNA进行富集, 并开发了全新的计算分析流程CIRCexplorer对环形RNA进行系统的预测分析, 在计算和实验水平确证了互补序列介导的外显子环化, 并揭示了基因组内互补序列的选择配对及其动态调控导致的可变环化现象(alternative circularization)[30]。
通过对高表达环形RNA进行的系统分析, 我们发现了一系列环形RNA生成的基因组结构特征。首先, 绝大多数形成环形RNA的外显子位于基因的中间位置, 而只有很少的外显子位于基因的两端, 这提示环形RNA的产生与RNA的剪接密切相关。因此, 我们在构建重组表达载体时充分考虑到了这一位置特征, 将产生环形RNA的序列加入到报告基因的中间区域来模拟环形RNA产生的天然条件。其次, 虽然大多数的环形RNA包含了两个及两个以上的外显子序列, 单一外显子序列形成的环形RNA只占到很少的一部分, 但是, 单一外显子形成的环形RNA其外显子长度相比于其他环形RNA的外显子更长, 这提示外显子反向剪接导致的环化需要一定的空间距离才能发生。最后, 我们也发现形成环形RNA外显子的上下游内含子序列显著地偏长, 并且富含同源重复Alu序列, 这与之前的报导一致[27]。但是进一步的分析发现, 这些Alu序列在环形RNA外显子上下游的长内含子中反向联排, 理论上可以通过反向互补Alu序列形成RNA分子内的折叠配对(IRAlu), 进而拉近反向剪接位点之间的距离, 促进外显子环化的发生。
我们进一步利用重组表达载体, 对反向互补Alu序列对于环形RNA产生的重要性开展实验验证和机制研究。由于形成环形RNA外显子的上下游内含子序列普遍偏长, 我们挑选了一个具有较短上下游内含子序列的环形RNA形成区域开展研究。其位于POLR2A基因内, 在上游内含子内有一个负向的Alu序列, 在下游内含子内有两个正向的Alu序列, 理论上可以形成两种不同组合的IRAlu配对。在进行的一系列Alu元件突变表达载体中, 当至少有一个IRAlu配对存在时(不论是IRAlu完全配对还是IRAlu部分配对), 我们都可以检测到重组环形RNA的存在。相应的, 当突变完全破坏所有的IRAlu配对时, 环形RNA的产生则很难被检测到。但是, 当我们将来源于不同基因上的IRAlu配对序列重新导入失活表达载体后, 尽管重组的效率可能有所差距, 环形RNA又可以重新产生。有意思的是, 我们构建了一个含有非重复互补配对序列的环形RNA高表达重组质粒, 表明环形RNA的形成需要两侧内含子区域的反向互补序列配对, 而这些反向互补序列可以是像Alu一样的重复序列, 也可以是非重复序列。进而, 我们也检测到了内源存在的非重复序列互补配对介导的环形RNA产生。有意思的是, 对不同物种转录组数据分析的结果提示, 互补序列及其介导的环形RNA产生在不同的物种间存在着动态差异。
通过大量的生物信息学分析和比较研究, 我们发现并不是所有的反向互补序列配对都可以导致环形RNA的产生, 即反向互补序列配对是环形RNA产生的必要但非充分条件。例如, 在人的ZWILCH基因中有一个环形RNA产生的潜在位点, 其上下游内含子区域间可以通过反向互补形成多个IRAlu配对, 但是我们并没有检测到ZWILCH位点环形RNA的表达; 然而在小鼠的ZWILCH基因内, 我们也预测到了相同位置上的多个反向互补配对(在小鼠中为SINE序列), 更为重要的是, 在该位点我们检测到了小鼠环形RNA的表达。那么究竟是什么原因导致了这种环形RNA表达的差异呢?我们提出了反向互补序列配对竞争导致环形RNA产生的理论。理论上来讲, 反向互补序列配对既可以发生在一个内含子内部序列中, 也可以发生在两个内含子间的序列中。当反向互补序列配对发生在一个内含子中时, 相邻外显子的剪接位点可以被拉近并通过正常的剪接产生线形RNA; 但是, 当反向互补序列配对发生在两个内含子中时, 位于两个内含子间的外显子则通过反向剪接发生环化作用, 形成环形RNA。这种反向互补序列的竞争性配对则导致了线形RNA和环形RNA形成的竞争性关系(图1A)。通过对整个人类基因组进行的系统分析发现, 反向互补序列的竞争性配对(在人中主要是IRAlu配对)不光与互补序列的绝对数目相关, 也与反向互补配对序列间的距离有关, 提示环形RNA有复杂的加工调控机制。有意思的是, 在我们工作发表的同一天, Mol Cell期刊上发表了另外一个环形RNA工作, 也从另一个角度揭示了线形RNA和环形RNA形成的竞争性关系[31]。
在研究中我们还惊奇地发现, 同一个基因区域可以产生多个环形RNA分子(图1B)。更为重要的是, 我们通过Northern blot进一步鉴定了这些环形RNA分子的存在。我们将这种一个基因前体RNA可以产生多个环形RNA的现象称为可变环化(alternative circularization), 对应于一个基因前体RNA产生多个(线形)mRNA的可变剪接(alternative splicing)。这种可变环化的现象也在多个人源细胞系的转录组数据中得到证实。我们推测, 可变环化的产生与人类基因组内含子区域中蕴含着的大量重复互补序列(主要是Alu)密切相关, 这些互补序列的选择配对及其动态调控使得同一个基因可以产生多个环形RNA分子。我们的这一发现也揭示了位于基因组内含子区域中Alu元件的一个新功能。
环形RNA研究展望
我们的此项研究成果首次证明了内含子RNA互补序列介导的外显子环化(环形RNA形成), 并提出了不同区域间互补序列的竞争性配对导致线形RNA或是环形RNA的产生; 重要的是, 这种互补序列的竞争性配对在不同物种间呈现差异的组合模式, 使得外显子来源环形RNA的表达具有物种特异性; 更为重要的是, 在人类基因组内含子区域中蕴含着大量的互补序列(如Alu等序列), 这些互补序列的选择配对及其动态调控导致了可变环化的发生[30]。这一系列的发现揭示了环形RNA这一类新型非编码RNA在体内的广泛存在, 进一步以全新的理论视角揭示了基因表达在转录/转录后水平的复杂性和多样性, 为深入研究外显子环化、可变环化以及与转录和剪接的协调作用机制奠定了坚实的分子基础。
这些已有的环形RNA研究丰富了我们对真核生物转录组复杂性的认识, 但是还有很多疑点有待破解[32]。例如, 这种反向剪接导致的外显子环化是如何与正常剪接相区分的?又是如何和转录/剪接协同作用的?其他影响环形RNA产生的顺式元件和反式因子是什么?可变环化是否受到空间结构的限制和调节?另外, 未来对环形RNA的研究将主要集中在其功能作用方面。尽管已有的一些研究结果表明, 内含子环形RNA可以通过结合RNA聚合酶II发挥顺式的转录调控作用[25]; 而个别的外显子环形RNA可以作为miRNA的分子海绵来发挥转录后调控作用[28,33], 但是大部分环形RNA的生物学功能及其作用机制不详。因此, 对环形RNA功能作用研究的突破, 将进一步推动我们对环形RNA的了解, 也会为我们带来更多的惊喜。
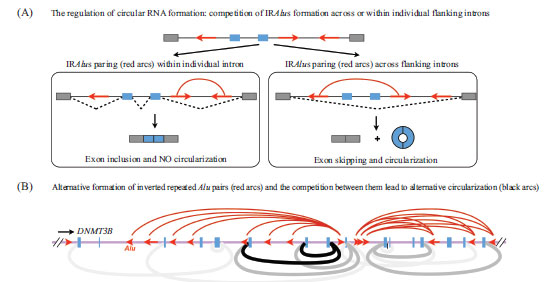
A: 内含子内的IRAlu的配对促进线性RNA的形成(左图), 内含子之间的IRAlu配对促进了环形RNA的形成(右图);
B: Alu之间不同的配对方式使得一个基因区域可以生成多个环形RNA, 称为可变环化。
A: the competition model of exon circularization. Left: the RNA pairing by IRAlus within one individual intron (red arrows) promotes normal splicing (dash lines), resulting in a linearized RNA transcript. Right: RNA pairing by IRAlus across flanking introns promotes back splicing, leading to exon circularization and a linearized RNA transcript with exon skipping.
B: alternative formation of IRAlus (red arcs) across introns and the competition between them lead to widely expressed alternative circularization (black arcs).
图1 外显子形成线性RNA和环形RNA的竞争性模型以及可变环化
Fig.1 The competition model of exon circularization and alternative circularization
文章来源|中国细胞生物学学报




来第一个抢占沙发评论吧!