随着转录组测序技术的成熟,科学家们发现circ-RNA并不是毫无技能的傻白甜,而是拥有特殊技能的百变大咖~于是乎,大家兴致盎然的想要一探究竟。可是,面对测序鉴定出海量的circRNA,小伙伴们往往陷入困境,这些circRNA是否真实存在?存在于哪里?到底发挥了什么功能?如何解决这些棘手的问题呢?不要方!本期小编就给各位介绍一下 circRNA的表达和功能验证方法。
circRNA的表达验证
circRNA qPCR验证
circRNA验证怎么做?毫无疑问,qPCR方法是首选,既简单又实用。
1.验证对象
表达量较高且表达差异较大的circRNA !!!选好对象真的很重要;
2.引物设计
别犯愁,一个图让你get到引物设计原则(图1)
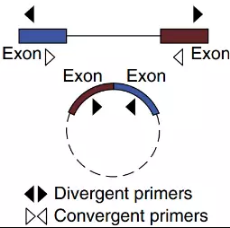
图1 circRNA引物设计示意图
(1) 线性RNA是根据目的片段的上下游分别设计引物,称之为convergent primer,扩增出来的片段就位于上下游引物之间(图1)。
(2) circRNA基于它的成环机制,除backsplice junction位点处不同于线性RNA之外,其他区域跟线性RNA基本都一致。circRNA验证时需针对backsplice junction位点处特异性的设计引物,称之为divergent primer(图1)。其他设计上的注意事项与常规PCR引物设计相同。此外,对于内含子环化的circRNA,除在backsplice junction位点处设计引物之外,还可以根据内含子区域设计引物。
3.验证策略:

图2 circRNA表达验证过程
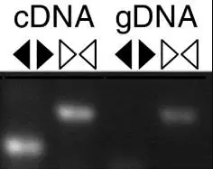
图3 circRNA表达验证电泳结果
circRNA定位
通过验证的circRNA究竟在细胞的哪个位置表达呢?正所谓“屁股决定脑袋,位置决定功能”。位于细胞核中的circRNA主要调控亲本基因的转录,而位于细胞质中的circRNA主要发挥竞争性内源RNA—ceRNA的作用 。因此,确定circRNA表达定位,更有利于我们后续确定对其功能研究的方向。一般采用FISH(fluorescence in situ hybridization)荧光原位杂交技术来进行定位分析(图4)。探针设计时需跨越backsplice junction位点区域。
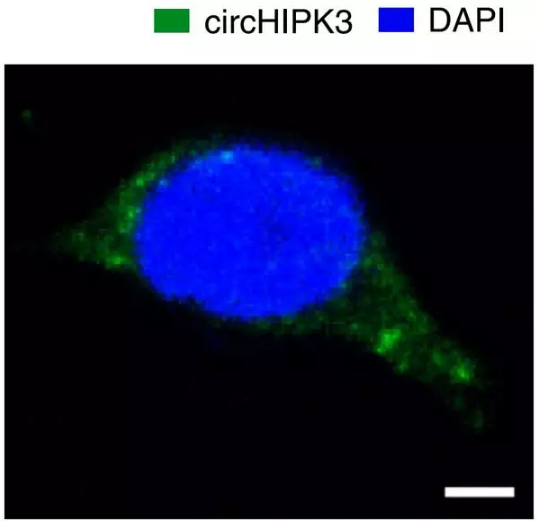
circRNA功能验证
circRNA过表达
侧翼反向互补序列诱导
目前较为普遍的做法是基于circRNA的形成机制,侧翼序列碱基互补配对——Alu结构(图5)介导的非线性剪接成环。基于此,PCR扩增circRNA外显子序列,包括侧翼Alu序列连接至表达载体中。将连接载体转染到相应的细胞样本中,定量PCR检测转染效率。然后用divergent primer验证circRNA过表达倍数。
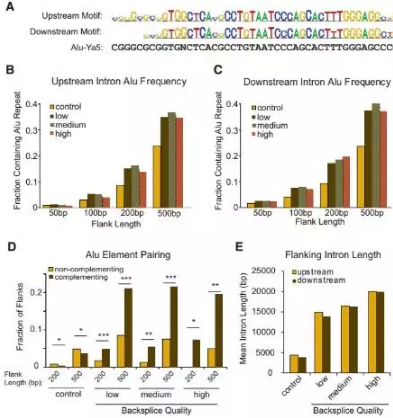
图5 circRNA侧翼结构特征
Tips:
1. 扩增目的区域包含circRNA侧翼Alu序列或内部碱基互补序列,扩增的侧翼上下游序列长度为1kb时表达效率较好;
2. 目的区域扩增时选用的是基因组DNA为模版。
tRNA内含子剪接形成
2016年有学者发表了一种基于tRNA改造的circRNA表达载体新技术(图6)。在前体tRNA加工的过程中,核酸内切酶TSEN会剪切掉tRNA基因内的内含子序列,随后通过RNA连接酶RtcB连接,形成成熟的tRNA。被剪切掉的内含子序列也通过RtcB酶作用头尾两端连接形成一个稳定的circRNA,称之为tricRNA。这个机制首先是在古生菌中被发现的,之后发现在后生动物中也非常保守。虽然这个tricRNA的形成并不依赖反向互补的序列,但tRNA内含子两侧的序列是高度保守的,且这段内含子序列必须具有bulge-helix-bulge(BHB)motif这样的结构特征。
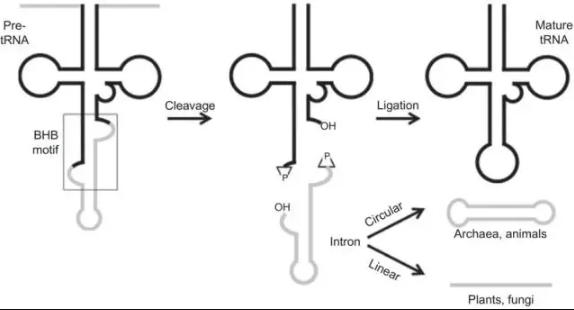
图6 tricRNA环化机制
构建策略:
1.克隆tRNA序列,在内含子BHB区域设计酶切位点;
2.在tRNA基因上游增加一段聚合酶III的启动子(5S、7SK、U6)序列。另外在完整启动子序列的基础上多增加一小段核酸序列能够提高启动子效率;
3.在两个酶切位点之间插入需要表达的circRNA序列(需保留部分BHB序列以保证BHB motif结构);
- 两种circRNA表达载体构建策略比较

大家根据实际情况按需选择,结合多种分析验证手段得出更加严谨的结论。
circRNA沉默
沉默circRNA,可利用siRNA进行干扰,针对cir-cRNA backsplice junction位点区域序列设计siRNA,对于内含子环化circRNA,也可针对内含子区域设计相应siRNA。同前文中介绍的方法一样,利用Divergent primer验证circRNA敲降倍数-敲降效率。需要注意的地方是设计siRNA时也需设计相应的对照,即backsplice junction位点一端序列互补配对,另一端则错配。(图7)。
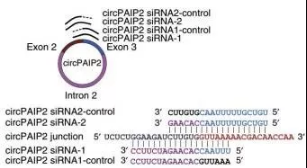
图7 circRNA沉默策略
小编就给大家介绍到这里,小伙伴们如有疑问欢迎您在下方评论,小编一定知无不言言无不尽!
参考文献
1. Li Z, Huang C, Bao C, et al. Exon-intron circular RNAs regulate transcription in the nucleus[J]. Nature Structural & Molecular Biology, 2015, 22(3):256.
2. Qu S, Yang X, Li X, et al. Circular RNA: A new star of noncoding RNAs[J]. Cancer Letters, 2015, 365(2):141-8.
3. Zheng Q, Bao C, Guo W, et al. Circular RNA profiling reveals an abundant circHIPK3 that regulates cell growth by sponging multiple miRNAs[J]. Nature Communications, 2016, 7(11215).
4. Jeck W R, Sorrentino J A, Wang K, et al. Circular RNAs are abundant, conserved, and associated with ALU repeats [J].RNA, 2012, 19(2):141-157.
5. Lu Z, Filonov G S, Noto J J, et al. Metazoan tRNA introns generate stable circular RNAs in vivo[J]. RNA, 2015, 21 (9):1554-65.
6. Schmidt C A, Noto J J, Filonov G S, et al. Chapter Nine-A Method for Expressing and Imaging Abundant, Stable, Circular RNAs In Vivo Using tRNA Splicing[J]. Methods in enzymology, 2016, 572: 215-236.
文章:美吉生物





好帖子,解决了我许多的疑惑;文笔严谨且透着些许幽默。
楼主优秀,楼主能写一篇设计CIRRNa引物和探针的文吗?期待哦
好帖子,很受用,感谢楼主。请教一下,一个母本基因在一种细胞系中有十几个circRNA,怎么选择做哪一个或哪几个?
根据重要性,序列长短,差异大小去选择