如题,今天我们来介绍Starbase:http://starbase.sysu.edu.cn/index.php


这个网站我们在(资源帖)S5E28: lncRNA常用数据库大盘点(上)提到过,但是没有深入展开,此网站甚是强大,当为研究非编码RNA必备神器之一。下面我们重点看最常用的四大功能:
一、根据microRNA找非编码RNA(比如lncRNA,circRNA等)
二、根据microRNA找mRNA靶点;
三、查找ceRNA调控分子;
四、查找RNA的结合蛋白;
下面我们直接看例子:
一、根据microRNA找非编码RNA
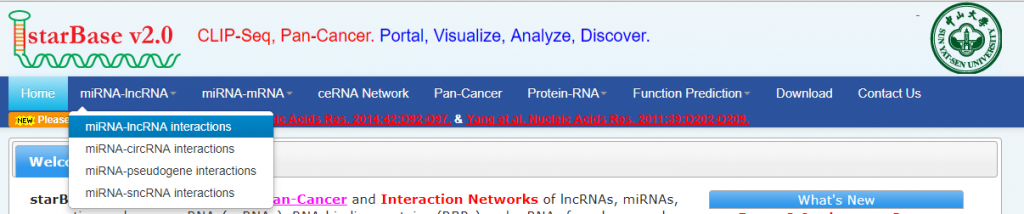

在上图选择miRNA-lncRNA互作,我们以miRNA 125a-5p为例,选择miRNA 125a-5p:
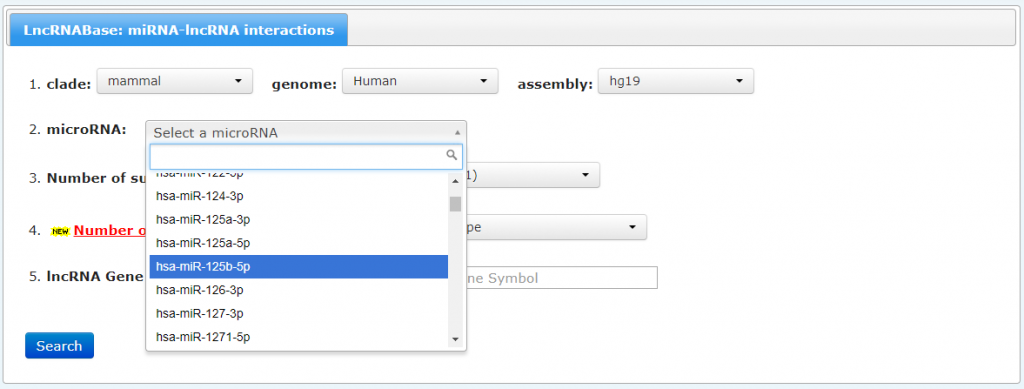

选择筛选的严格程度, 我们按照最低的:low stringency:
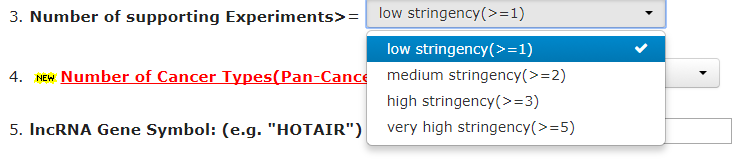

接下来:
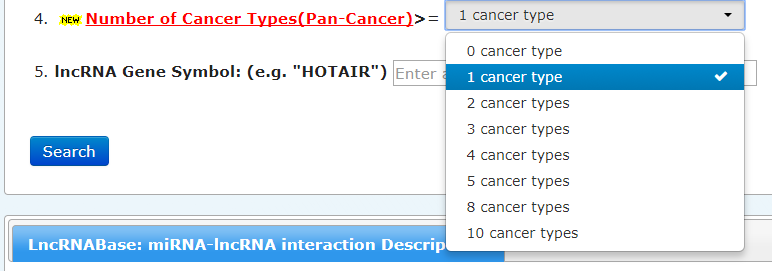

选择在14种肿瘤中,至少出现1个,lncRNA可以空着:
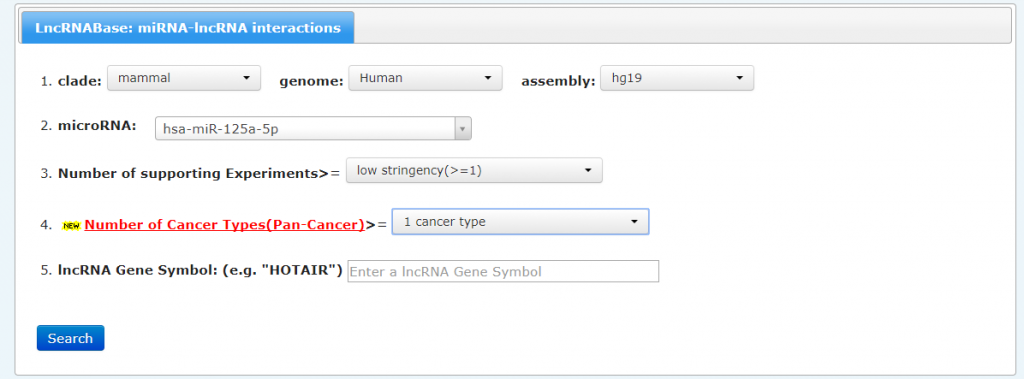

点击Search,出现两条:
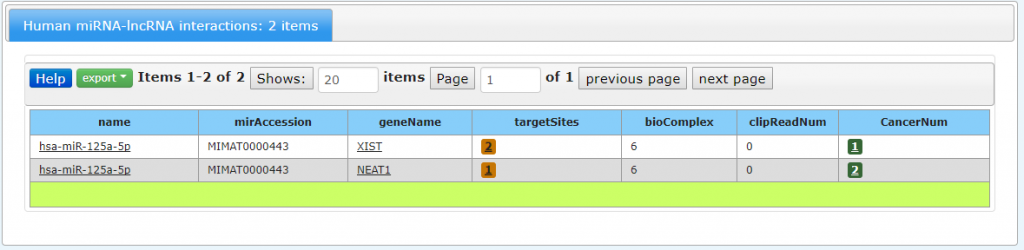
一个是Targetsite,
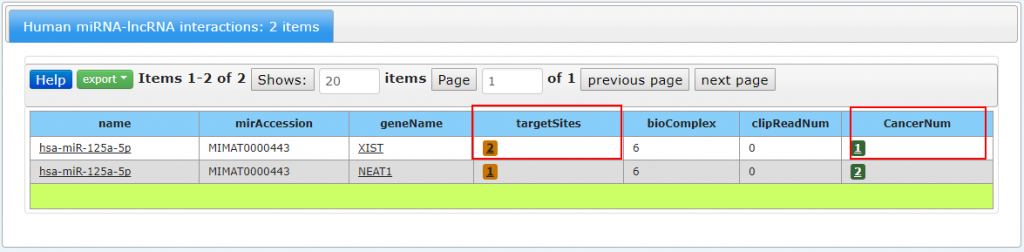
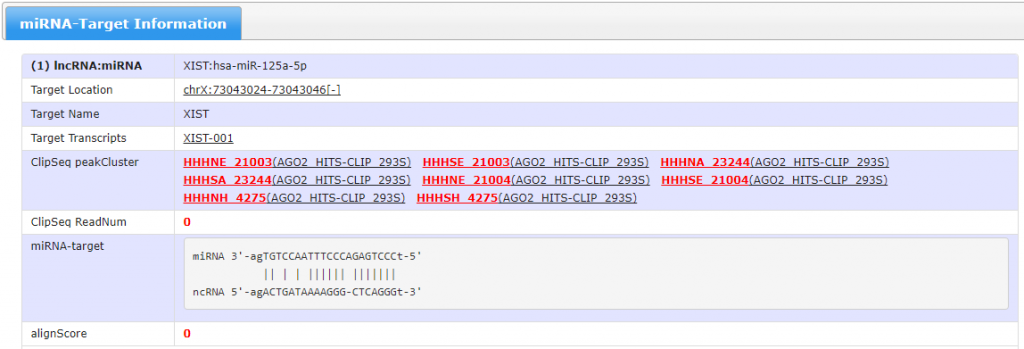
单击显示具体的结果:
然后选择Cancer Num:
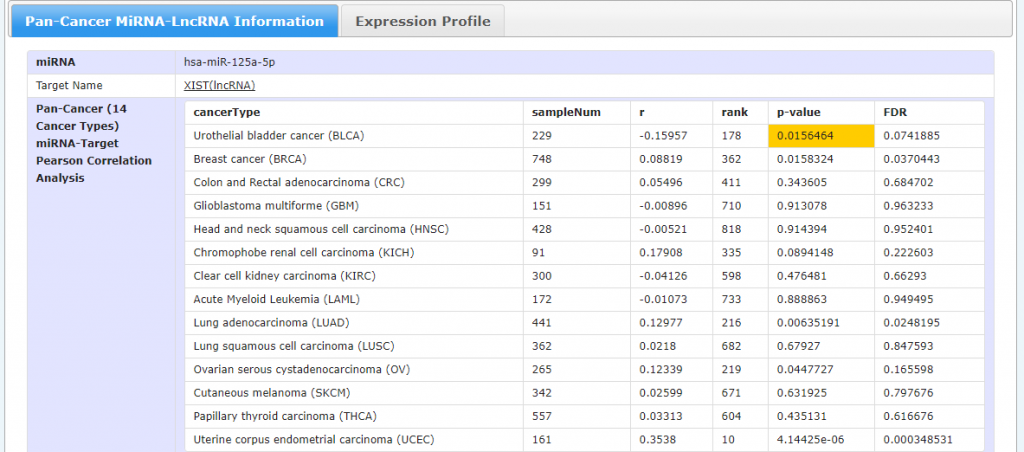
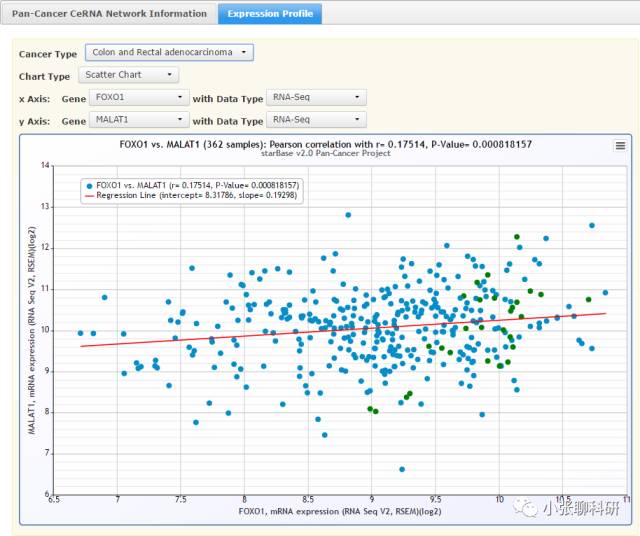
这里显示的是hsa-miR-125a-5p和XIST(lncRNA)表达负相关,r=-0.159,P=0.0156。接着点击Expression Profile:
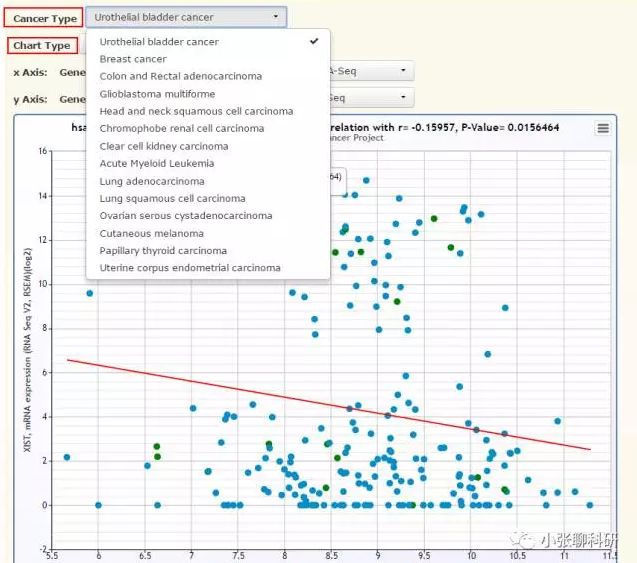
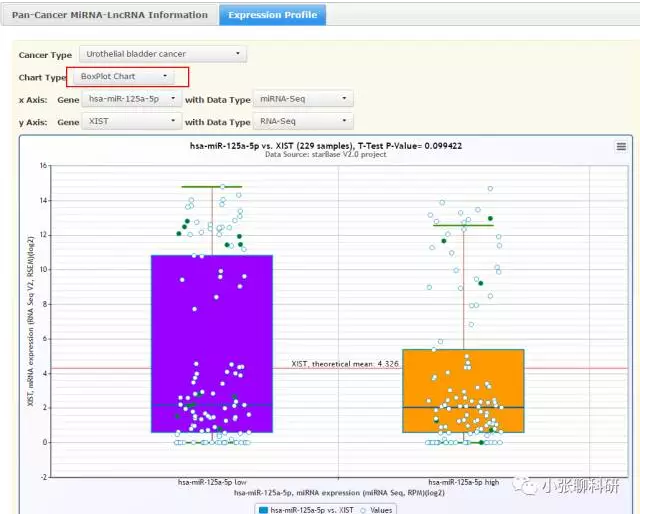
选择Cancer Type肿瘤类型和Chart Type图标类型,这里我们选择Boxplot箱式图:
当然,如果大家觉得只有Xist和NEAT1两条lncRNA,数量少些,那可以把肿瘤数量设置为0:
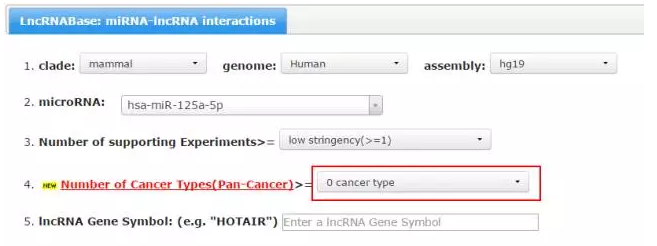
这样出来的lncRNA就多了,出现了27条lncRNA:
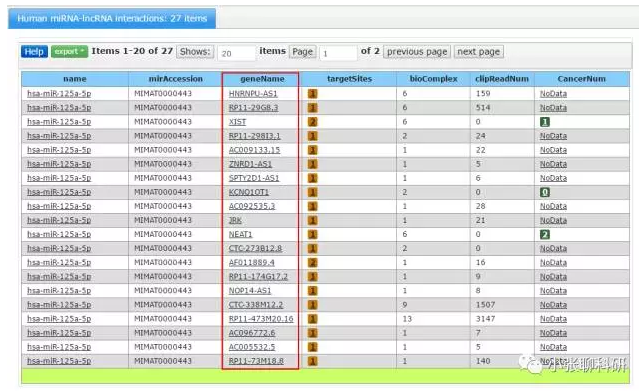
二、根据microRNA找mRNA靶点
我们选择miRNA-mRNA interaction,同样的设置:
预测到954个mRNA:
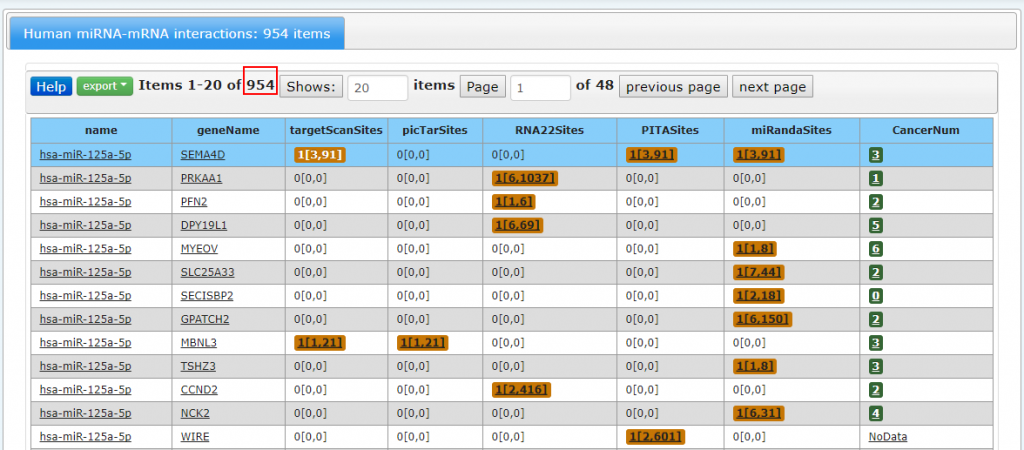
我们看到这里的预测软件是Pictar,RNA22,PITA和MiRanda,单击第一个基因SEMA4D的CancerNum也可以看到结果:
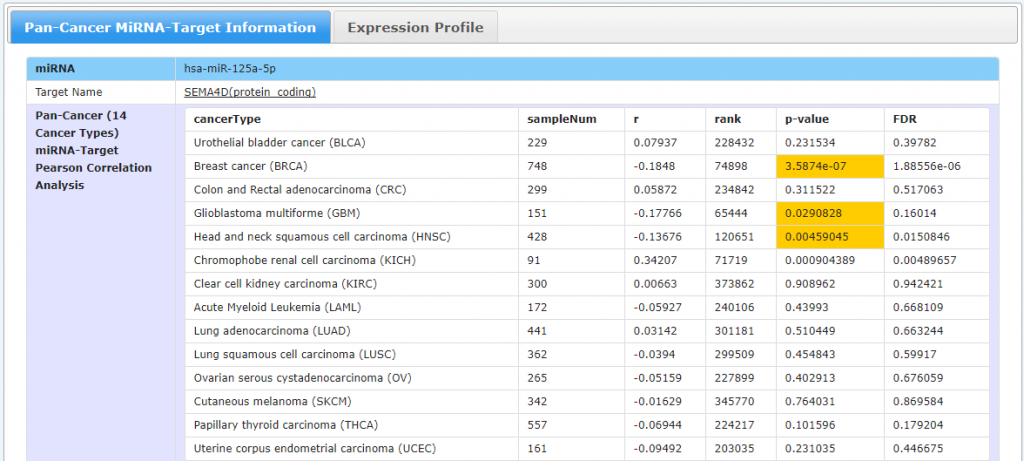
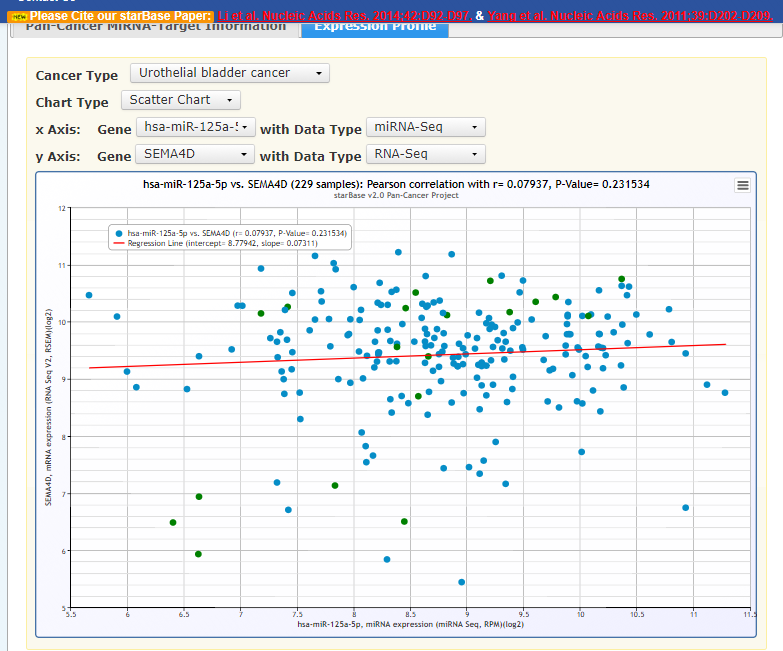
同样也可以看到microRNA和mRNA的表达相关性:
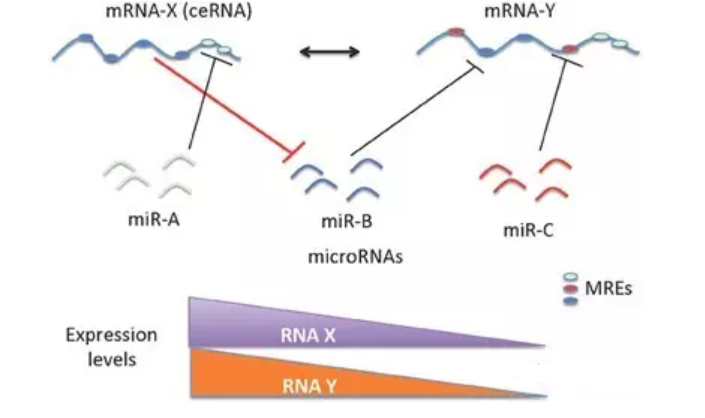
三、查找ceRNA调控分子;
ceRNA的作用机制我们以前介绍过:聊科研系列•第三期: 竞争性内源RNA(敌之敌,即吾之友)
在ceRNA network中,我们以FOXO1为例:
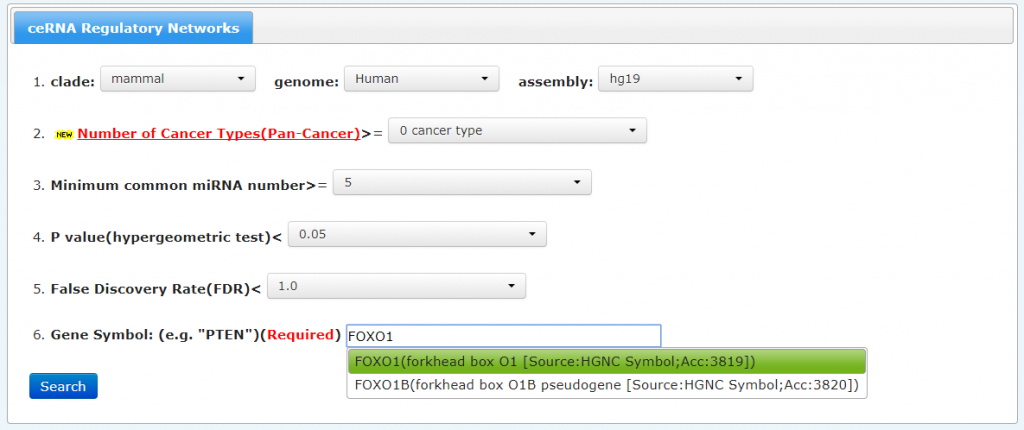
然后搜索:
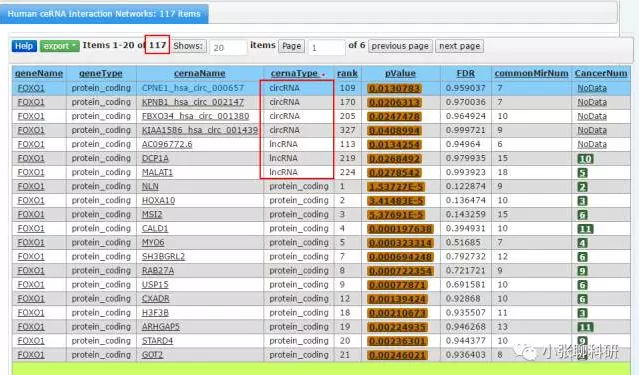
单击ceRNA type,我们看到有三条circRNA和3条lncRNA:
我们分别单击MALAT1的P value和CancerNum,两者有18条共同的miRNA:
肿瘤数量跟我们前面看过的一样:
四、查找RNA的结合蛋白;
我们选择第一项protein-lncRNA interactions,可以选择结合蛋白:
也可以直接输入lncRNA:
结果就出来了:
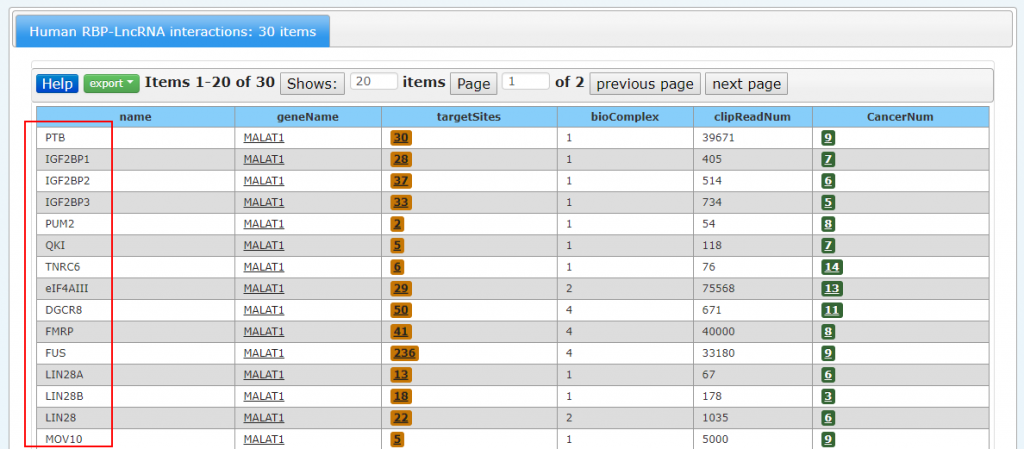
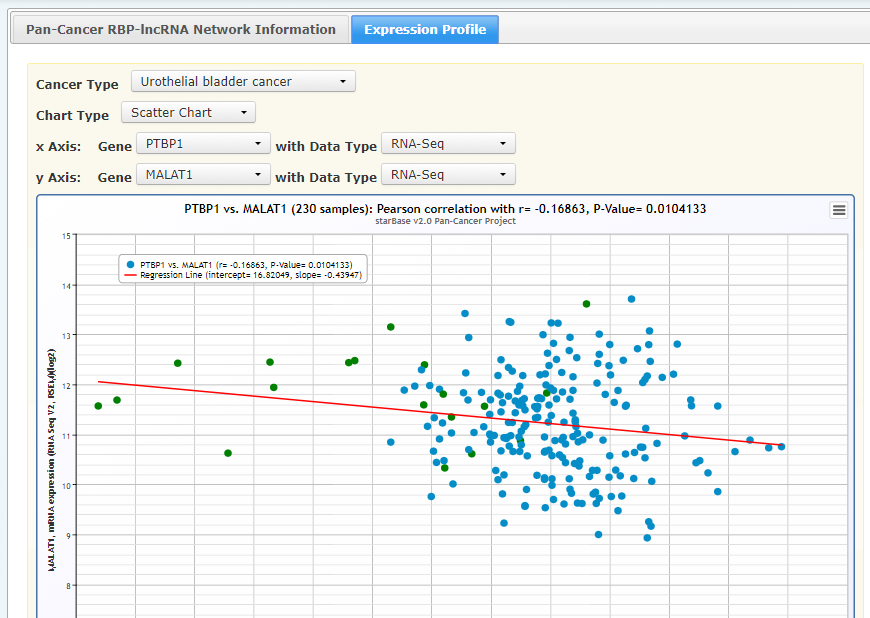
单击CancerNum确定蛋白与lncRNA表达相关性:
好了,今天的内容就到这里了。
文章:小张聊科研



谢谢LZ的分享,我按照LZ的方法试的时候,为什么点expression profile后没有出来图而是提示error:Your request could not be completed.?是数据库的问题吗?
starbase v3.0 可以显示expression profile
starbase可以从circRNA预测可能结合的miRNA吗?