Identification of circular RNAs and their alterations involved in developing male Xenopus laevis chronically exposed to atrazine
期刊:Chemosphere 影响因子:4.208分
发表单位:山东省职业卫生与职业病防治研究院
发育期雄性蟾蜍长期暴露于阿特拉津(除草剂),睾丸组织circRNA的鉴定及其差异表达分析
1 摘要
阿特拉津(Atrazine,简称AZ)是一种外源激素,可影响两栖动物正常生长发育。作者以往研究发现,AZ剂量100ug/L会引起发育期雄性蟾蜍发育异常,引起与性腺相关基因表达改变。最近,有人发现circRNA在多种发育异常的组织中参与作用。但是否有circRNAs参与AZ处理过的蟾蜍并不了解。本研究,通过对对照组(n=3)和AZ处理过的蟾蜍(n=3)的睾丸组织进行测序,鉴定到68575个circRNAs。AZ处理过组织中405个circRNAs表达有差异,其中44个上调,361个下调。通过qPCR对2个上调,6个下调的circRNAs进行验证,发现结果与转录组测序结果一致。282差异表达circRNAs发挥miRNA海绵吸附作用。KEGG通路分析发现,miRNAs靶基因主要涉及到19个pathway,其中Wnt通路, 孕激素介导的卵细胞成熟通路(progesterone-mediated oocyte maturation)可能是很重要的通路,本研究第一次提供证据显示,AZ能够影响雄性蟾蜍的正常发育,并能够调控睾丸中相关的基因表达。
2 研究方法
2.1 样品准备
NF分期47(卵孵化后13d), 来源于相同亲本,混合性别的蝌蚪,分成不同组,每个组(n=160),分成8个小组。AZ用二甲亚砜(DMSO, 0.01%, Sigma, St. Louis., MO, USA)溶解,处理组暴露于AZ(pu rity of 97%, Sigma, St. Louis., MO, USA)180天,对照组蝌蚪,用0.01% DMSO处理。
按照(Sai et al., 2016)方法,收集发育中雄性蟾蜍的睾丸,三个对照,三个处理,使用Trizol(Invitrogen, Carlsbad, CA, USA)提取TotalRNA,采用NanoDrop® ND-2000 光谱仪评估RNA的浓度和纯度,变性凝胶电泳检测RNA的完整性。
使用去除核糖体RNA的试剂盒TruSeq Stranded Total RNA Library Prep Kit(Illumina, SanDiego, CA, USA) 构建文库。BioAnalyzer 2100 system (Agilent Technologies, Santa Clara, CA, USA)评估文库的质量。文库解链成单链DNA分子,测序仪流动槽捕获测,PCR反应成簇,Illumina HiSeq 4000 进行150个循环反应生成PE150序列。
下机成对的序列,采用Q30控制质量,使用cutadapt软件去除3’接头及低质量reads。Clean reads将用于circRNAs分析,使用Bowtie2软件将序列比对到参考基因组/转录组(NCBI xl_ref_Xenopus_laevis_v2),使用find_circ软件鉴定circRNAs。成环剪切点reads,count值使用整体mapped reads进行校正,并进行log2转换。t-test进行两组差异表达分析,fold changes ≥2.0,P-values ≤0.05的circRNAs作为差异表达的circRNAs.
2.3 靶基因预测及生信分析
使用Targetscan和Miranda 软件对circRNAs 结合miRNA位点,及miRNA靶基因进行预测,并对差异表达的circRNAs相关的靶基因进行KEGG富集分析。
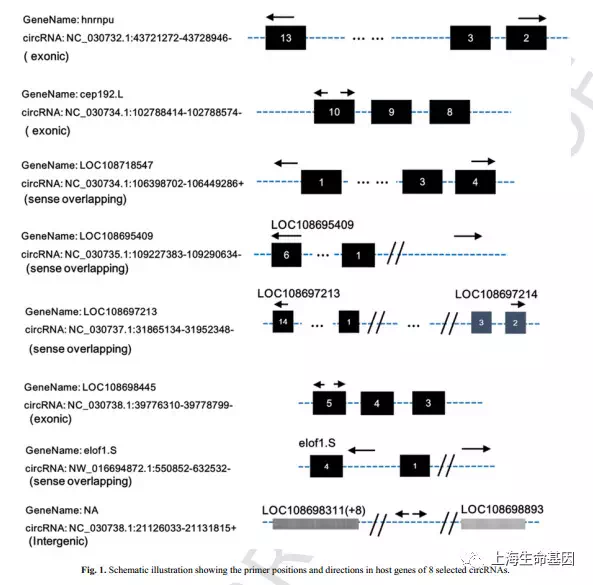
2.4 circRNAs验证
使用qPCR(Q-RT-PCR, ViiA 7 Real-time PCR System, Applied Biosystems, Carlsbad, CA, USA)对挑选的8个circRNA,进行验证,totalRNA 用 RNase R酶,消化线性RNA,引物采用divergent primer设计如Fig1,circRNAs的选择参考其潜在的功能和倍数两个因素。qPCR时,用Invitrogen Superscript cDNA Synthesis kit(Invitrogen Corp., Carlsbad, CA, USA)将TotalRNA转化成cDNA。RNase R酶处理时,TotalRNA(2ug)一部分用RNase R(20 U/μl, Epicentre)处理,一部分不用RNase R处理,37 °C 20min,使用2-△△CT法,计算表达量。2-△△CT>1时上调,2-△△CT<1下调,基因GPI用于内参。qPCR采用三重复结果以 mean±standard deviation形式呈现。使用SPSS Statistics 18.0 software(IBM Corp., New York, NY, USA)进行统计分析,使用student t-test at进行两组比较,p<0.05认为有差异。
3 研究结果
3.1 蟾蜍circRNAs鉴定
对照组三个样品Clean reads分别得到85457078、85154262、70110988条,匹配到参考基因组的reads数分别是45908497、44827626 、37089055。经过AZ处理的处理组得到的Clean reads数目是89071634、95827312、84718576,匹配到参考基因组的reads数目是46499156、52443972、41544671。一共鉴定到68575 个circRNAs。
3.2 差异表达分析
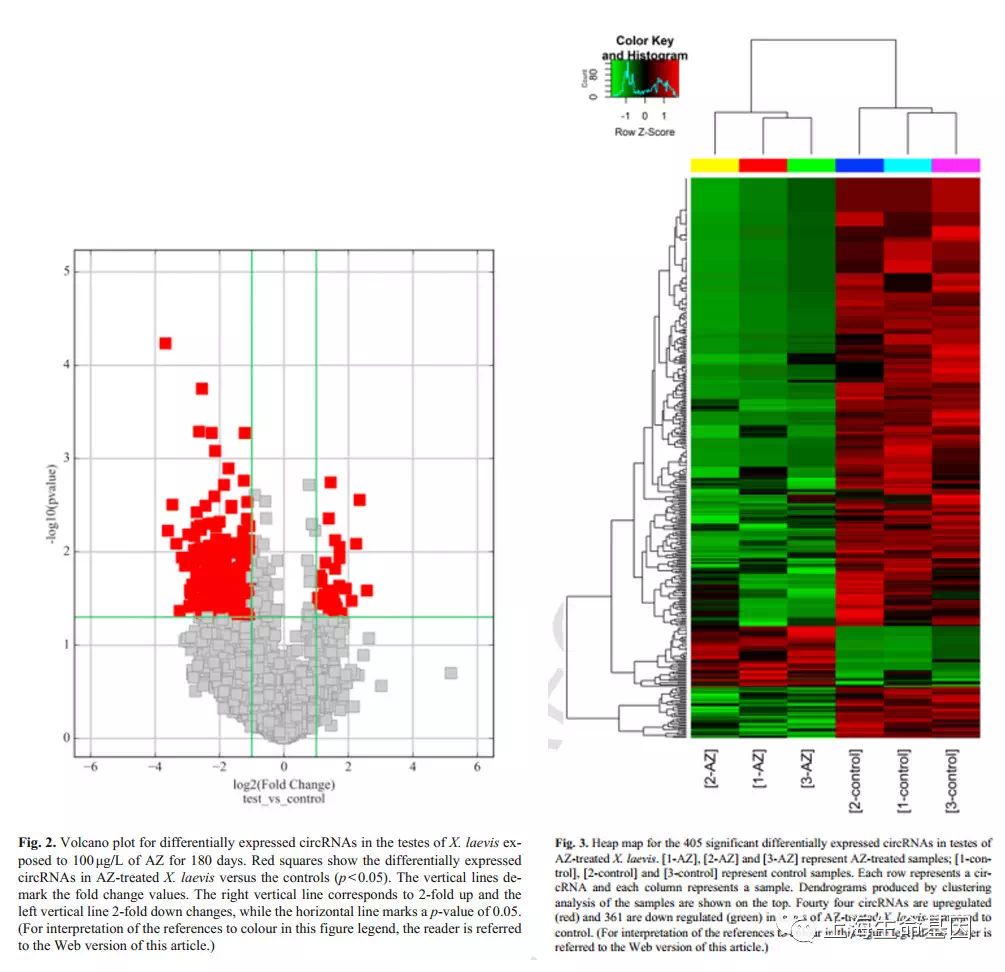
处理组与对照组相比,共有405个circRNAs表达有差异,44个上调,361个下调,火山图如Fig.2。热图展示差异表达的circRNAs如Fig.3,行代表单个circRNAs,列代表样品名字,上面的树图代表样品聚类,左边的部分是AZ处理组,右边是对照组。本研究原始数据存储在GEO数据库中,编号为GSE103526。
3.3 circRNAs的miRNA结合位点预测,并构建circRNA-miRNA网络
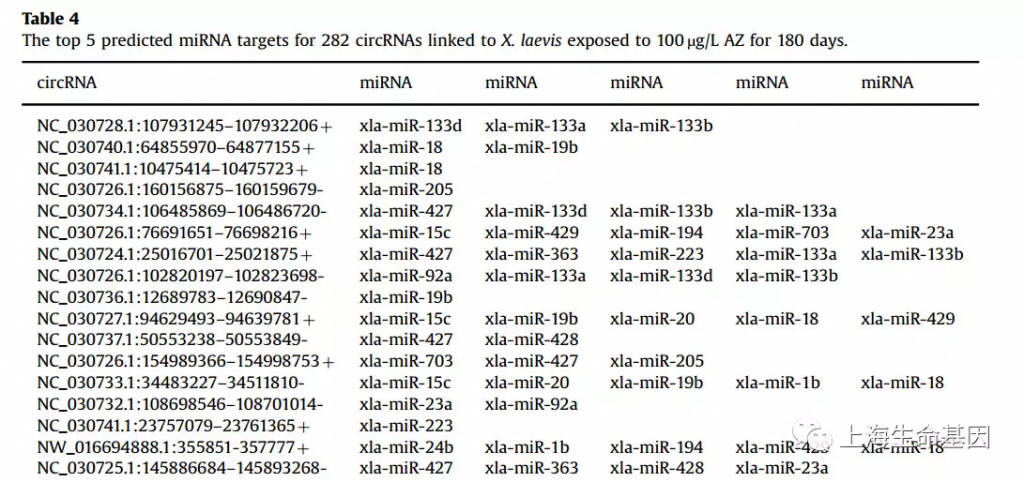
对差异表达的circRNAs进行miRNA预测,发现282个circRNAs中含有miRNA结合位点,结合位点数量从1-21不等。对这282个circRNAs选取前5个结合位点最多的miRNA, 如Table 4。然后自由选择了5个circRNAs,分别是NC_030735.1:109227383-109290634-, NC_030724.1:105611901-105637401+, NC_030734.1:106398702-106449286+, NW_016694872.1:550852-632532-, NC_030735.1:34874500-34913276-。构建circRNA-miRNA网络。如 fig4。
3.4 circRNA相关靶基因KEGG富集分析
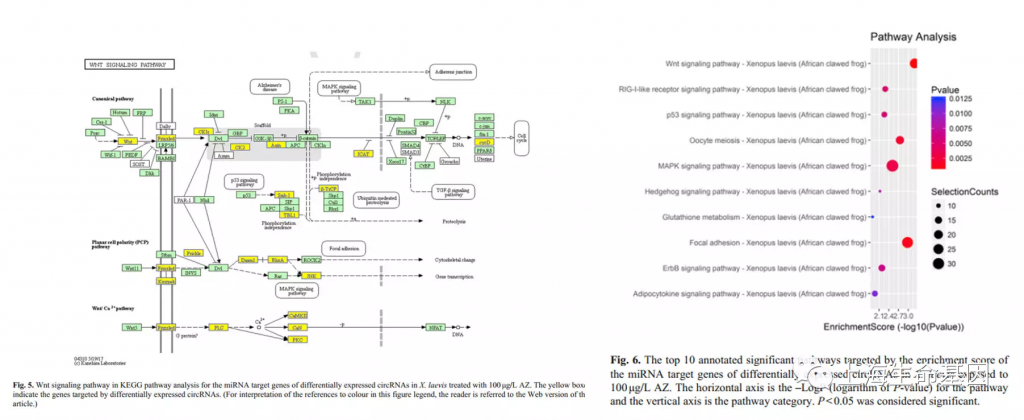
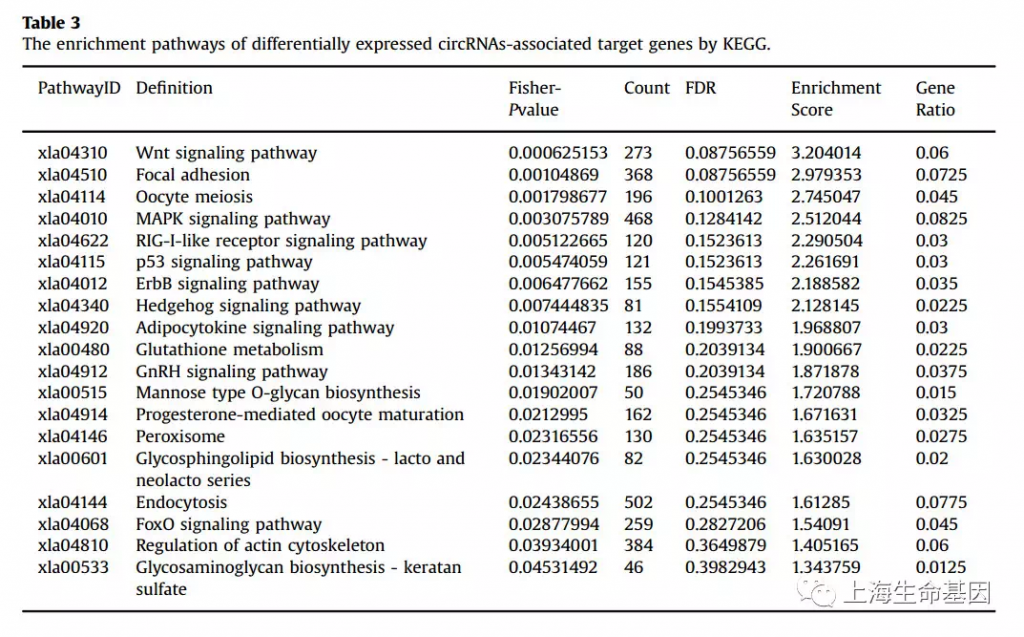
基于circRNAs相关的靶基因进行KEGG富集分析,发现19个信号通路,Table3, 其中,enrichment score排序发现,Wnt信号通路排在最前,并包含21个miRNAs靶基因,Wnt通路图如Fig.5。前十个通路展示如Fig.6。

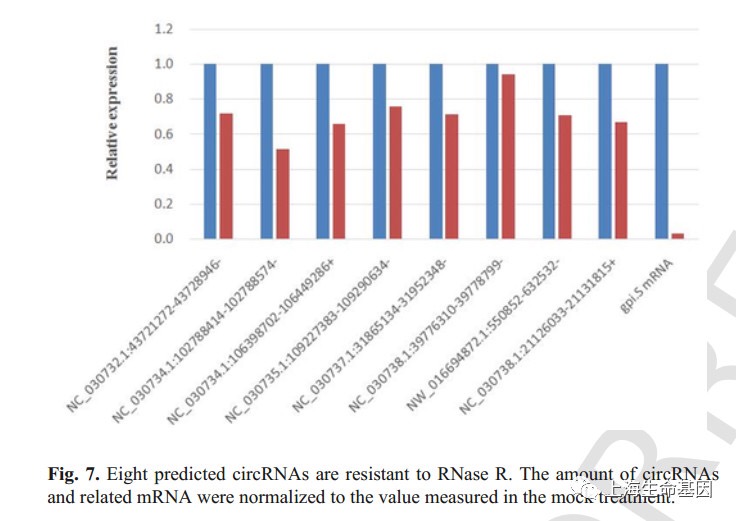
3.5 circRNA验证
对8个circRNAs进行qPCR发现,2个circRNAs是上调的(NC_030738.1:39776310-39778799- 和NC_030738.1:21126033-21131815+),6个circRNAs是下调的(NC_030734.1:102788414-102788574-, NW_016694872.1:550852-632532-,
NC_030737.1:31865134-31952348-, NC_030735.1:109227383-109290634-, NC_030734.1:106398702-106449286 + and NC_030732.1:43721272-43728946-;)qPCR验证的结果与circRNA测序的结果趋势一致。RNase R酶消化实验显示,这些RNA确是环状的。如图Fig7.
4 讨论
AZ是一个常用的除草剂,对生态系统有一定的危害。暴露在AZ的环境,可能是两栖动物种群数量下降的一个重要原因,AZ能够影响他们的生长,代谢,免疫系统以及血细胞过程(Langerveld et al., 2009)。我们之前的研究显示AZ的毒性与蟾蜍基因水平的改变相关。AZ对蟾蜍的生存产生影响,而且睾丸的体重和性腺指数均显著下降。另外,AZ的不同剂量(0.01, 1, 10, 100μg/L)均会引起睾丸的退化,尤其是发育期的小蟾蜍暴露在100μg/L时。结果表明AZ通过干扰相关基因的表达量改变,影响雄性发育期蟾蜍的生殖能力和免疫系统。此结果也促使我们去研究其背后更多的潜在分子机制。
本研究,我们用qPCR验证了RNase R酶处理下,circRNA的表达,结果显示我们的方法有效的鉴定到了蟾蜍睾丸中circRNAs的种类。
circRNAs是一类近些年发现的一类非编码RNA,并受到广泛关注。circRNAs表达丰富,稳定,并且进化保守(笔者对circRNAs基因组来源序列多物种保守性分析发现确实很保守,circRNA大多来源于外显子剪切形成,外显子是基因编码区,不同物种间保守程度很高),在胞内分子调控网络和表观遗传中通过吸附miRNAs起重要的调控作用。Memczak et al. 发现,circRNAs在不同的组织和发育时期的表达不同。自从基于对基因组测序的方式发现circRNAs以来, 大量的circRNAs被报道出来,包括人,古细菌,线虫,小鼠等(Salzman et al., 2012; Danan et al., 2012; Memczak et al., 2013)。但,在两栖动物中还没有通过RNAseq技术报道过circRNAs,本研究,通过RNAseq,我们发现了68575个circRNAs。之前没有关于AZ处理蟾蜍睾丸组织中circRNAs的数据。本研究第一次提供证据,表明circRNAs在受到AZ处理的蟾蜍睾丸组织中有差异表达。之前有报道称circRNAs的表达量变化与疾病的发生有关(Lin et al., 2016),因而,我们可以推断,circRNAs的改变与蟾蜍睾丸退化有关。
Hansen et al. 报道circRNAs的吸附作用是一个普通的现象。circRNAs通过与miRNAs的结合,调控miRNAs的靶基因。我们发现405个差异表达的circRNAs中有282个circRNAs有miRNAs结合位点,数量1-21个不等。蟾蜍中circRNAs作为miRNAs海绵吸附作用与之前在人、鸡、小麦等物种中报道的类似(Memczak et al., 2013; Zhang et al., 2016; Wang et al., 2016)。我们之前的研究发现基因表达量改变在AZ处理的蟾蜍睾丸退化中发挥重要作用。因此,我们推断这些改变的circRNAs通过miRNA海绵吸附作用,阻止miRNAs与其调控的靶基因结合,进而在发育异常组织中发挥作用。
我们进一步分析了circRNAs的靶基因的功能,在KEGG通路中共注释到19个显著的通路,并发现在Wnt通路中有21个靶基因(mRNA).Wnt通路会受到胞内多种信号分子的调控,进而引起多种细胞响应,包括基因转录,增殖,分化,迁移等(Kikuchi et al., 2009; Grumolato et al., 2010; Simons et al., 2012),Wnt通路的精确调控在正常发育和组织维持中是极其重要的(Schneider et al., Rudloff and Kemler, 2012)。因而,我们推测,AZ处理的蟾蜍睾丸退化中,与Wnt信号通路相关的差异circRNAs起到了很重要的作用。
以往研究,在差异表达的基因中我们发现了6个信号通路。孕酮介导的卵母细胞成熟信号通路(Progesterone-mediated oocyte maturation pathway)就是其中一个。有趣的是,此通路在本研究中也被鉴定到。因而我们推断,该通路也与AZ处理蟾蜍睾丸异常发育有关。
这些数据同样给出了一个启示,差异表达的circRNAs可能可以作为新的雄性生殖毒理的分子标志物。而且,本研究为进一步研究AZ处理蟾蜍睾丸退化中circRNA调控分子机制,提供了一个坚实的基础。并且,本研究有助于我们理解circRNAs的一般功能,以及在环境如AZ响应中的作用。
5 结论
本研究,在蟾蜍睾丸组织中鉴定到68575个circRNAs。 circRNAs的表达谱显示circRNA的表达量改变与睾丸生殖毒理密切相关。405个circRNAs差异表达,其中44个上调,361个下调。282个差异表达的 circRNAs发挥了miRNA海绵吸附作用。KEGG通路显示,这些circRNAs主要涉及到19个通路,其中Wnt通路,Progesterone-mediated oocyte maturation通路是两个非常重要 的通路。我们的研究有助于理解,AZ处理的雄性蟾蜍生殖毒理中潜在的的分子机制。
本研究思路简要整理:
研究目的:雄性蟾蜍睾丸退化中circRNAs是否参与该过程
实验方法:
材料:AZ处理的发育期雄性蟾蜍睾丸及未经AZ处理的对照,测序样品为3对3设计。
RNAseq测序策略:做circRNA测序通常有两种建库方式,一种是去核糖体rRNA,RNase R去线性RNA,链特异性建库的方式;另一种是去核糖体rRNA, 不去线性RNA,而对所有长链RNA,进行链特异性建库的方式。本研究采用第二种方式,没有去处线性RNA。采用HiSeq 4000上机,PE150测序模式。
circRNA差异分析:先使用find_circ对clean reads进行处理,获得circRNA count值,再对count值,使用total mapped reads进行校正,并做log2转换。然后使用t-test方法进行差异分析。fold changes ≥2.0,P-values ≤0.05的circRNAs作为差异表达的 circRNAs.
海绵吸附作用miRNA预测:使用Targetscan 和Miranda 软件预测circRNA结合的miRNA(没有miRNA的表达量数据,此miRNA会用该物种所有miRNA进行扫描),由于一个circRNA可能会与几十,几百个miRNA结合,此处选择前5个结合位点最多的miRNA,作为circRNA的结合miRNA(此处确定miRNA的目的是为了进行构建,circ-miRNA网络及miRNA靶基因预测)。
靶基因富集分析:circRNA-associated target genes富集,通常有两类,一是circRNA的来源基因,另一类是circRNA 结合的miRNA,miRNA的靶基因。作者对circRNA潜在结合的miRNA进行了预测,并做了miRNA 的靶基因预测。KEGG分析,基于miRNA的靶基因。
circRNA qPCR验证:挑选8个进行qPCR验证,采用RNase R处理的和没有处理的两种方式。405个有差异的,circRNA选择哪些呢,作者考虑circRNA的表达量变化差异(差异比较大),及circRNA的重要性(此处考虑circRNA 结合miRNAs进而调控的重要基因,选择重要的circRNA)。
文章结果展示:
1. 对下机数据clean reads及mapped reads进行统计描述。
2. 差异表达的circRNA进行了统计描述,并用火山图和热图展示。
3. Circ-miRNA结合,选取了5个circRNA(可以结合miRNA的circRNA有282个,如果都用,这个网会非常的大),构建circ-miRNA网络。
4. KEGG对circRNA 结合miRNA 的靶基因进行富集分析,富集到19个通路,附件表格列出19个通路的信息,正文用散点图展示top10个通路,作者重点关注 Wnt通路,并在文中放了Wnt通路图,标注颜色为miRNA的靶基因。
5. circRNA的验证,qPCR. 用柱状图说明,circRNA鉴定的准确性。




Mark,获益匪浅!