|
Circular RNA expression profile analysis of severe acne by RNA-seq and bioinformatics
RNAseq技术和生物信息学分析重度痤疮circRNA表达谱
期刊:JEADV 影响因子:3.528
发表单位:广州医科大学
摘要
背景:痤疮是一种常见的慢性皮肤病,致病因素及发病机理多样。近些年,人们发现circRNAs在多种生物学过程和人类疾病中以circRNA-miRNA-mRNA的方式调控基因表达。但在痤疮病人中circRNAs的表达谱并未报道过。
目的:研究重度痤疮circRNA 表达谱。
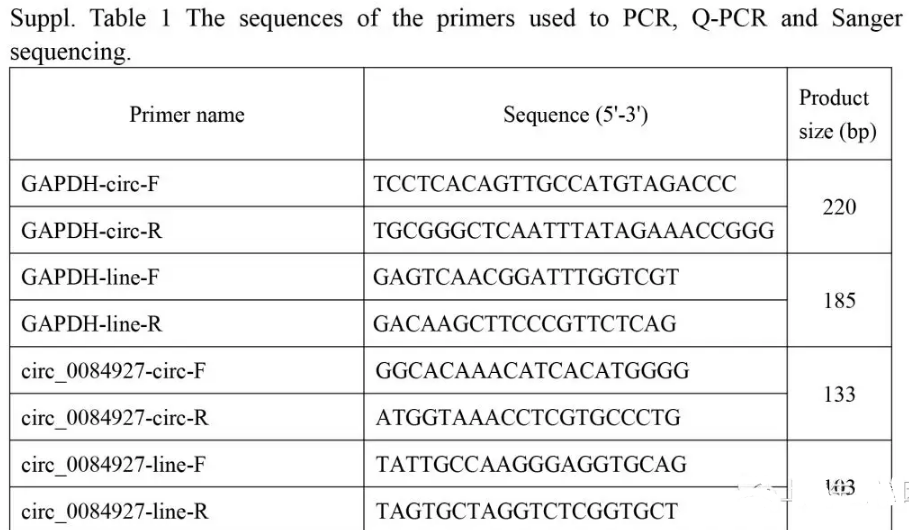
方法:通过高通量RNAseq技术和生物信息学方法研究三对重度痤疮病损皮肤及周围非病损皮肤组织。RT-PCR,Sanger测序,qPCR(别的样品n=4),验证后续circRNAs。预测circRNA-miRNA-mRNA关系网络。
结果:重度痤疮相比周边正常组织,病损组织中有538个circRNAs差异表达,其中271个上调,267个下调。GO,KEGG富集分析发现异常表达的circRNAs主要涉及到inflammatory(炎症), metabolism(代谢), and immune responses(免疫应答)。RT-PCR,Sanger测序,qPCR验证5个候选circRNAs(circRNA_0084927, circRNA_0001073, circRNA_0005941, circRNA_0086376, and circRNA_0018168),均显著下调,结果与RNAseq结果一致。此5个circRNAs可以与213个miRNAs结合,并调控靶基因表达。
结论:本研究第一次揭示circRNAs在重度痤疮差异表达,circRNAs可以作为药物治疗痤疮的潜在标志物。
2 材料方法
1 材料
共收集7个病人(RNAseq分析:3男性,平均年龄22.3;PCR,Sanger测序,qPCR:4男性,平均年龄:21),至少两个皮肤病学家依据Pillsbury分级系统准确判断为重度痤疮(grade IV)。取病人配对的病损组织及旁边非病损组织(病损区2cm),本研究获得广州皮肤病研究所皮肤科医学伦理委员会批准,并遵从Helsinki Guidelines声明进行。
2 Illumina 测序
Trizol (Invitrogen, Carlsbad, CA, USA)提取total RNA。RNaseR酶消化线性RNA,RNAClean beads纯化,剩余RNA二价阳离子适宜温度片段化。随机六聚体引物合成第一链cDNA。AMPure 磁珠纯化片段,溶于EB buffer,用于末端修复并在3’端添加A,而后添加接头构建cDNA文库。最后,文库质检合格后,Illumina HiSeq 4000 platform 测序,模式为双端100bp。
3 circRNAs鉴定
下机数据质控后,将clean reads先比对到参考基因组(GRCh37/hg19),参数为bwa mem -T 19。CIRI预测circRNAs,gencodeV19. annotation.gtf 用于注释文件。为了与circBase数据库比较,circRNAs使用TPM校正,并使用Pearson比较每个样本的相关性。为了研究circRNAs保守性,使用phyloP软件计算circRNAs序列的保守得分,平均得分。
4 circRNA差异表达分析
分别用NOISeq and PossionDis进行差异表达分析。过滤掉表达量极低的circRNAs,NOISeq probability > 0.8并且PossionDis FDR< 0.001认为是有显著表达差异的circRNAs。对候选基因进行GO,KEGG富集分析,并基于STRING构建蛋白互作网络。
5 circRNA验证
特殊的circRNAs引物扩增total RNA和基因组DNA。PCR产物2% (w/v)琼脂糖凝胶分离,Sanger测序。引物如TableS1。GoTaq® qPCR Master Mix (A6002, Promega, USA) qPCR分析。熔解曲线分析引物特异性,2−△△Ct计算circRNAs相对差异表达。
6 预测circRNA-miRNA-mRNA关系
使用TargetScan和miRanda两种算法(基于miRBase 21.0)预测circRNAs结合的miRNAs。基于miRTarBase数据库预测miRNA-mRNA关系。Cytoscape构建circRNA-miRNA-mRNA关系网络。
7 统计分析
Wilcoxon rank-sum test, Student’s t-test and fold change 计算测序数据circRNAs的差异。qPCR结果计算Pearson’s相关性。P < 0.05认为有意义。差异表达circRNAs和mRNA的差异表达倍数设置为≥2.0。
3 结果
1 circRNA表达谱
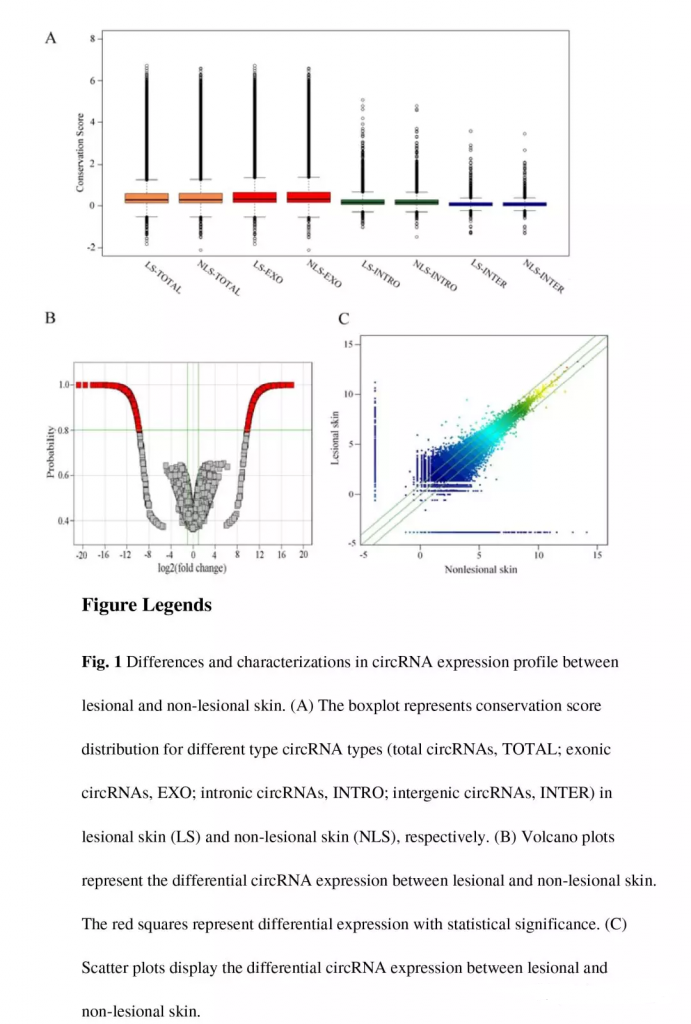
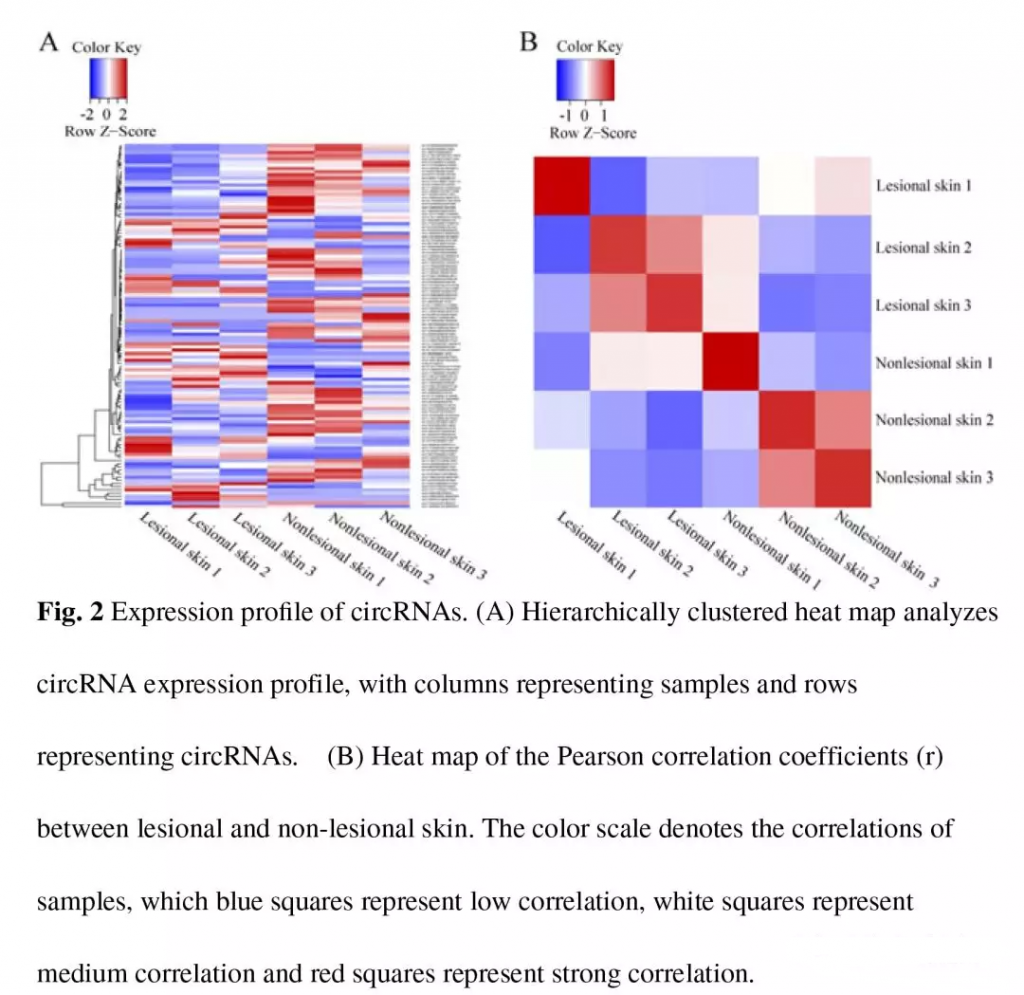
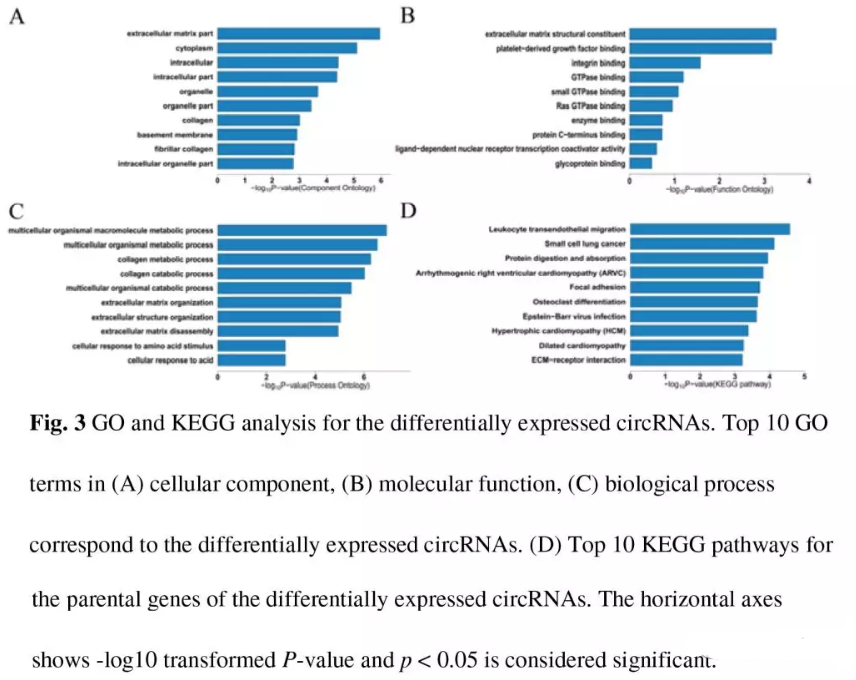
病损皮肤中共鉴定到35782个circRNAs,非病损皮肤中共鉴定到27700个。两者circRNAs的类型(全部circRNAs,外显子circRNAs,内含子circRNAs,基因间circRNAs)的分布基本相似(Fig. 1A)。大部分的circRNAs(病损皮肤87%,非病损皮肤84%)来源于蛋白编码基因外显子,剩余其它则来源于内含子或基因间区域。两组间circRNAs的表达差异用三点图评估(Fig.1B),circRNAs差异表达火山图(Fig.1C)。差异表达circRNAs热图(Fig. 2A),不同分组间circRNAs表达的相关性(Fig. 2B).。共鉴定到538个差异的circRNAs,271个上调,267个下调。其中330个是新的circRNAs(不存在与circBase数据库),比如(cLCE2B, cAHNAK, and cANKRD36BP2)。分析差异表达circRNAs富集到的top10 GO (cellular component, molecular function and biological process)。KEGG富集分析发现主要涉及到leukocyte transendothelial migration(白细胞跨内皮迁移), small cell lung cancer(非小细胞肺癌), protein digestion and absorption(蛋白消化和吸收), arrhythmogenic right ventricular cardiomyopathy(致心律失常性右室心肌病), and focal adhesion(粘着斑)(Fig. 3D)。
2 差异表达 circRNAs验证
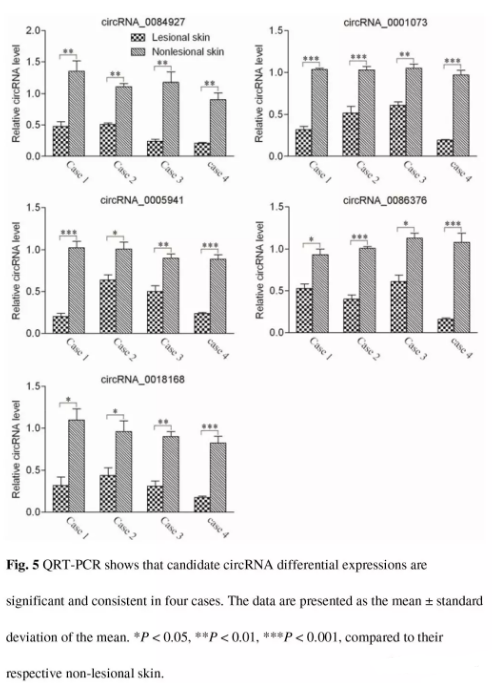
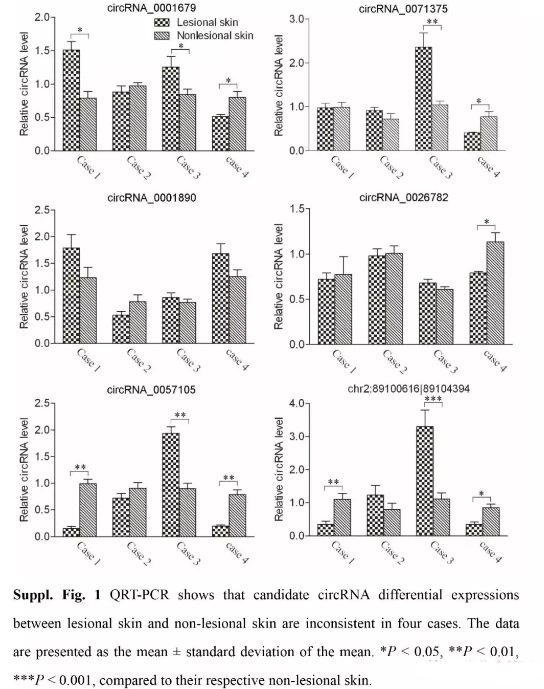
挑选15个显著差异的circRNAs用于验证,其中5个是新的(Table 1)。为了验证选择的是否确定是circRNAs,使用发散性divergent引物,和收敛convergent引物,分别进行RT-PCR。其中11个毫无疑问可以用发散性引物从cDNA中扩增出(Fig. 4A),剩下的4个不能,说明这些circRNAs的表达量很低或者没有表达。对于挑选得到的11个circRNAs使用Sanger测序对PCR扩增产物进行验证,鉴定heat-to-tail连接点(Fig. 4B)。这11个circRNAs进一步用qPCR进行验证,结果显示5个显著下调(Fig.5)与测序结果一致,而其他6个与测序结果不一致或没有显著差异变化(Suppl. Fig. 1)。


3 预测circRNA-miRNA-mRNA网络
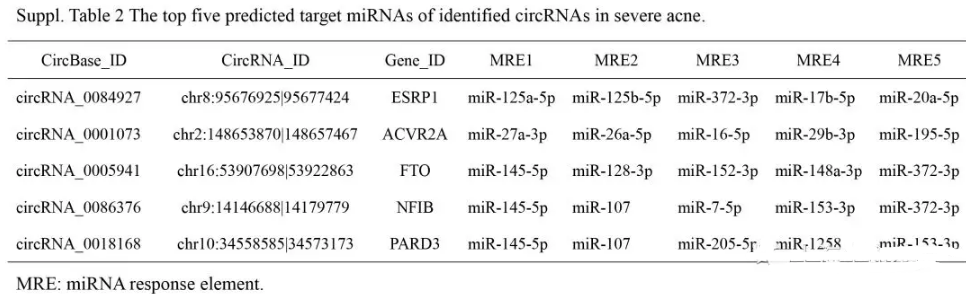
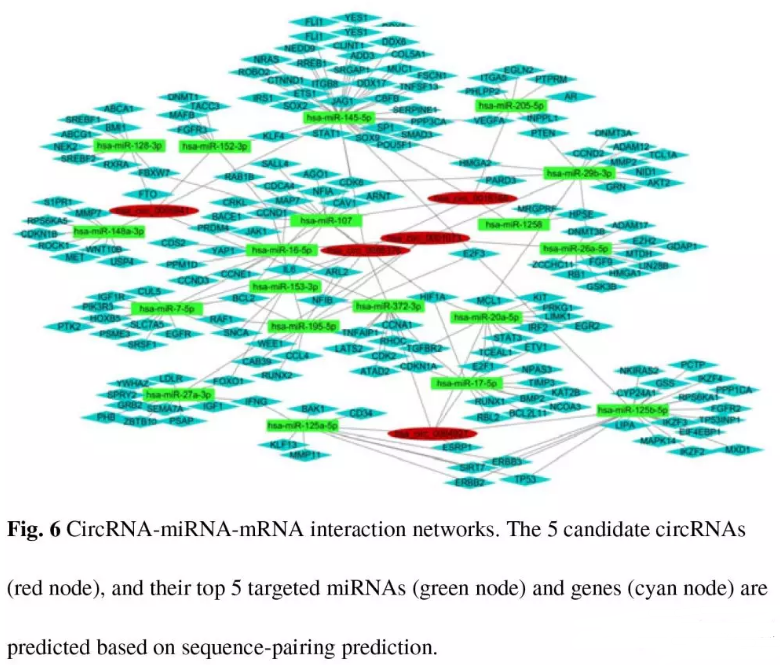
5个鉴定到的circRNAs可以预测到213个miRNAs。取前5个预测的miRNAs及其靶基因基因使用Cytoscape进行网络图展示(Fig. 6 and Suppl. Table 2)。其中部分circRNAs预测的miRNAs是重叠的,如miR-145-5p可以被3个circRNAs结合(circRNA_0005941, circRNA_0086376 and circRNA_0018168),是网络中最大的关系节点。
4 讨论
circRNAs是一种连续的共价闭环的分子,病毒,植物,动物中均有检测到。越来越多的研究者开始关注circRNAs的潜在功能,比如miRNAs海绵吸附调控基因的表达进而影响生理过程或疾病。最近有些miRNAs如as miR-143,miR-338-3p在调控痤疮的炎症反应中有重要作用。据我们所知,本研究第一次深入研究重度痤疮circRNAs表达谱及其潜在功能。使用高通量测序及生信分析,共鉴定到538个差异表达的circRNAs,其中271个上调circRNAs,267个下调。GO,KEGG富集分析显示差异表达的circRNAs主要与inflammatory, metabolism, and immune responses相关,这些在痤疮病理生理学中很重要。挑选11个circRNAs进行验证,5 circRNAs (circRNA_0084927, circRNA_0001073, circRNA_0005941, circRNA_0086376, circRNA_0018168)用于后续PCR, Sanger sequencing, and qRT-PCR验证,这些与测序结果一致,可能参与重度痤疮的起始与进展。然而,未来需要更大样本量及基于circRNA-miRNA-mRNA网络的发病机制研究。这些结果给我们研究重度痤疮机制提供了一个新的角度。
5 研究思路
材料:
7个病人(RNAseq分析:3男性;PCR,Sanger测序,qPCR:4男性)
研究方法:
1 建库采用去线性RNA,去核糖体RNA建库方式,HiSeq 4000,PE100测序。
2 CIRI预测circRNAs,gencodeV19. annotation.gtf 用于注释文件。circRNAs使用TPM校正,并使用Pearson比较样本相关性。phyloP软件计算circRNAs序列的保守得分,平均得分。
3 NOISeq and PossionDis进行差异表达分析。过滤掉表达量极低的circRNAs,NOISeq probability > 0.8 并且 PossionDis FDR< 0.001认为是有显著表达差异的circRNAs。对候选基因进行GO,KEGG富集分析,基于STRING构建蛋白互作网络。
4 PCR, Sanger sequencing, and qRT-PCR circRNAs验证。
5 TargetScan,miRanda,miRTarBase预测circRNA-miRNA-mRNA关系,Cytoscape构建网络图。
结果展示:
1 circRNAs表达谱: 保守性得分箱型图(Fig. 1A),两分组整体表达散点图(Fig.1B),差异表达火山图(Fig.1C),差异表达circRNAs热图(Fig. 2A),样品相关性图(Fig. 2B),GO,KEGG富集结果柱状图(Fig. 3)。
2 circRNAs验证:挑选15个circRNAs(Table 1),RT-PCR扩增电泳图(Fig. 4A),Sanger测序(Fig. 4B),qPCR验证(Fig.5,Suppl. Fig. 1)。
3 circRNA-miRNA-mRNA网络(Fig. 6 and Suppl. Table 2)。





来第一个抢占沙发评论吧!