转载:小张聊科研|微信公众号
我们以前介绍过一些研究circRNA常用的数据库和工具:circRNA数据库再更新,这次不只有肿瘤了!和如何设计circRNA的引物并检测其特异性?(含中文操作教程),今天我们继续介绍一个数据库:CircInteractome(Cirular RNA interactome):https://circinteractome.nia.nih.gov/index.html
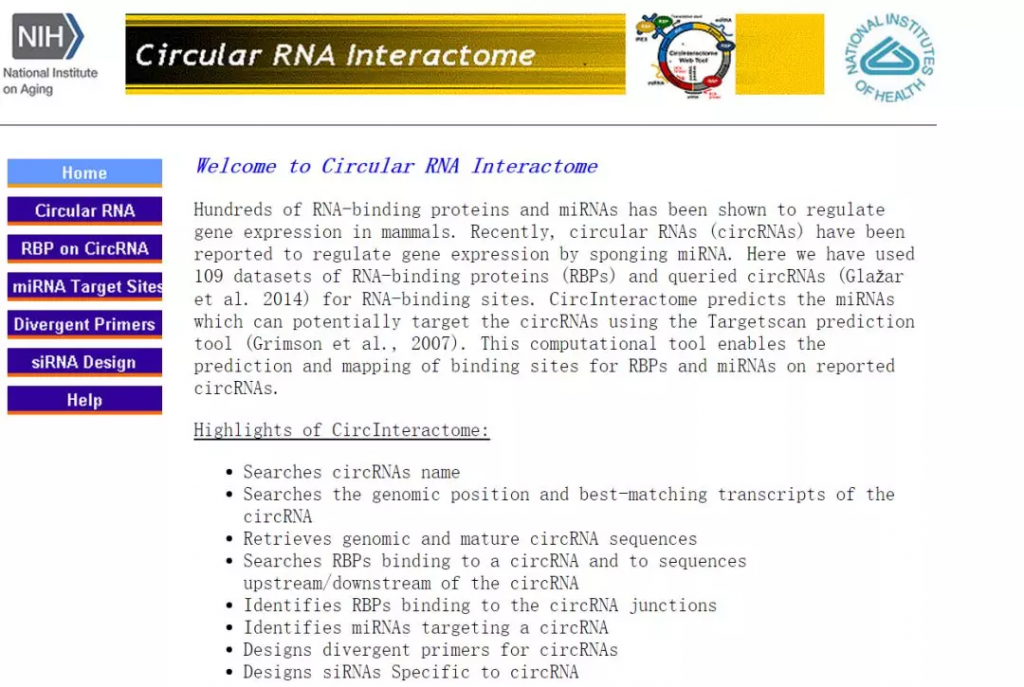
如上图所示,大家可以通过这个数据库
1、搜索circRNA的名字、序列、在基因组上的位置及结合的蛋白;
2、搜索circRNA结合的miRNA;
3、设计circRNA的引物及siRNA序列;
可以说功能很强大了。下面我们就具体来看看这些功能的实现。
一、搜索circRNA的名字、序列、在基因组上的位置及结合的蛋白
首先是circRNA的搜索:
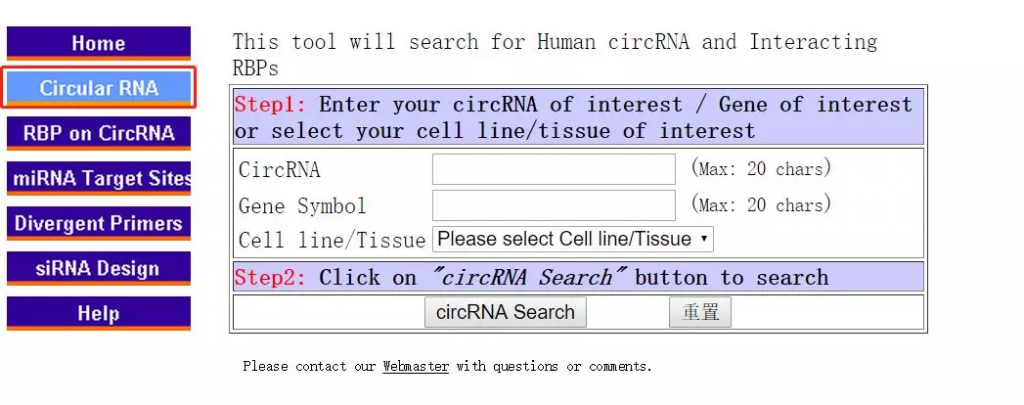
我们可以根据circRNA或者基因名进行搜索,比如以基因Cul2为例,在Gene Symbol后面的对话框中输入Cul2,然后单击circRNA Search:
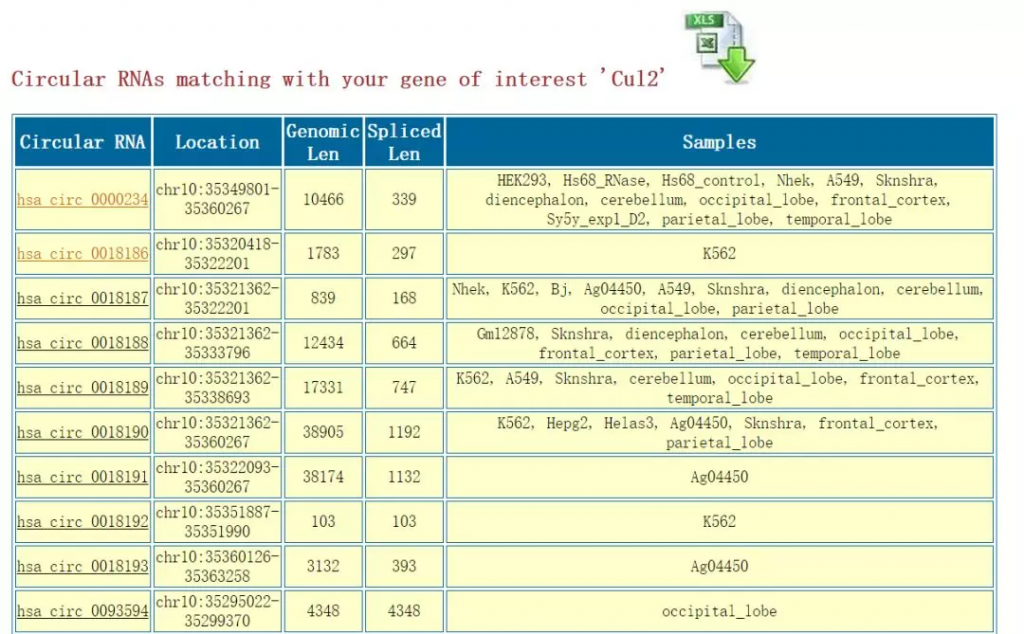
在新页面中就出现了这个基因来源的各个circRNA,基因组的位置、长度及在什么样本中表达,单击XLS图标还可以下载:
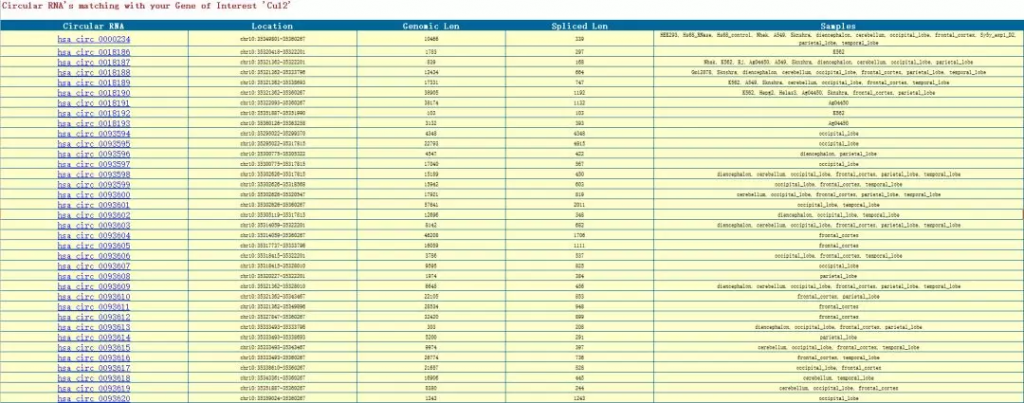
我们单击第一个hsa_circ_0000234后可以看到更加详细的信息:
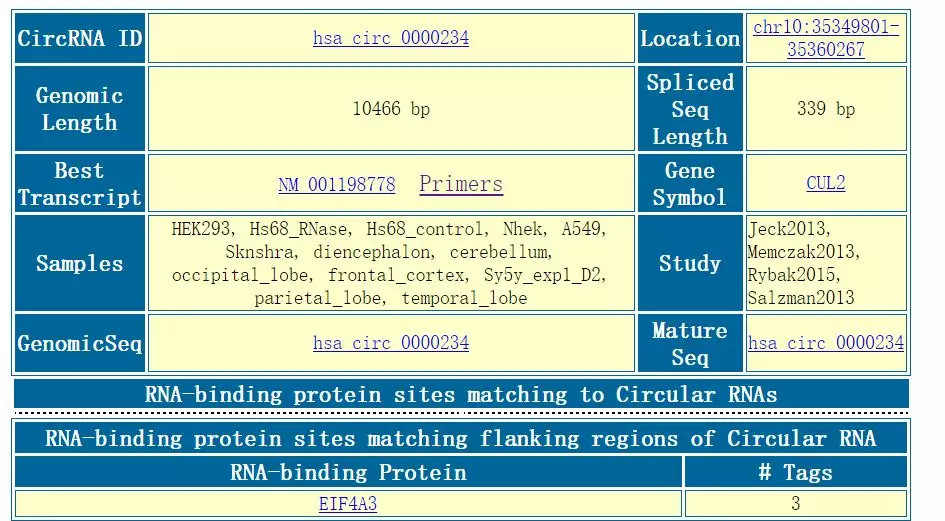
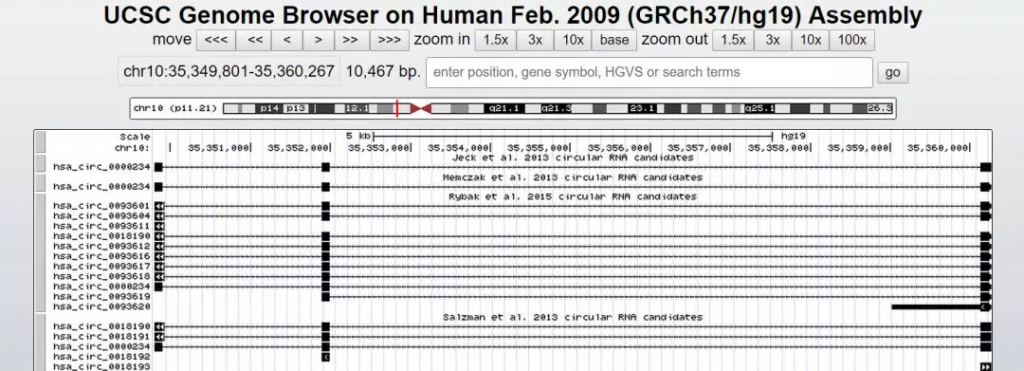
通过单击location可以在UCSC中查看位置:
通过单击Mature Seq可以看到circRNA的序列:

我们也可以看到预测到的与hsa_circ_0000234结合的RNA结合蛋白是EIF4A3。
当然,如果要反过来操作,根据RNA结合蛋白(RBP)来预测与之结合的circRNA:
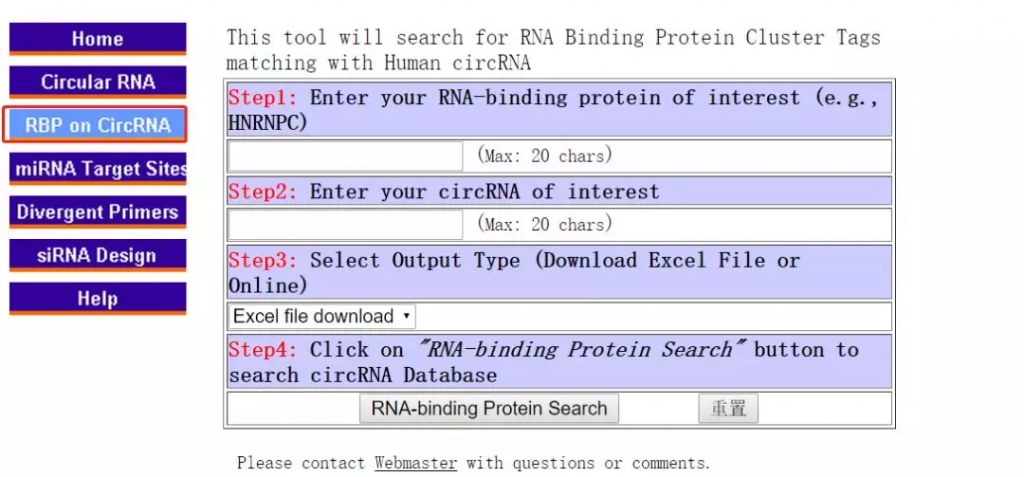
在右侧的界面中,我们可以输入RBP或者circRNA的名字,比如RBP我们用HNRNPC,在输出output type里面可以选择excel file download也可以选择web:
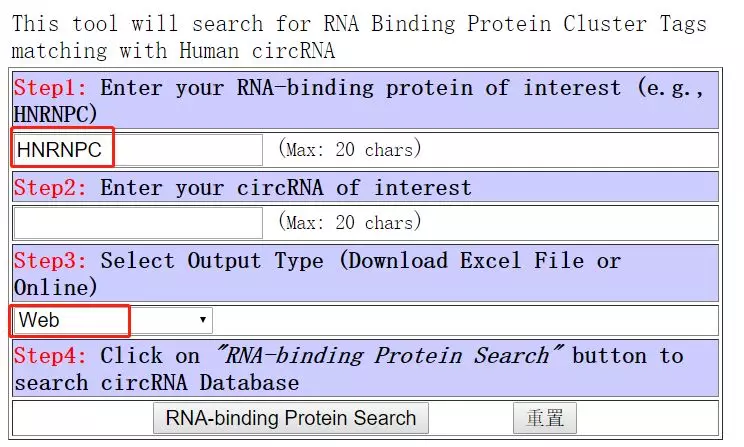
这样可以看到预测到的与HNRNPC结合的circRNA:
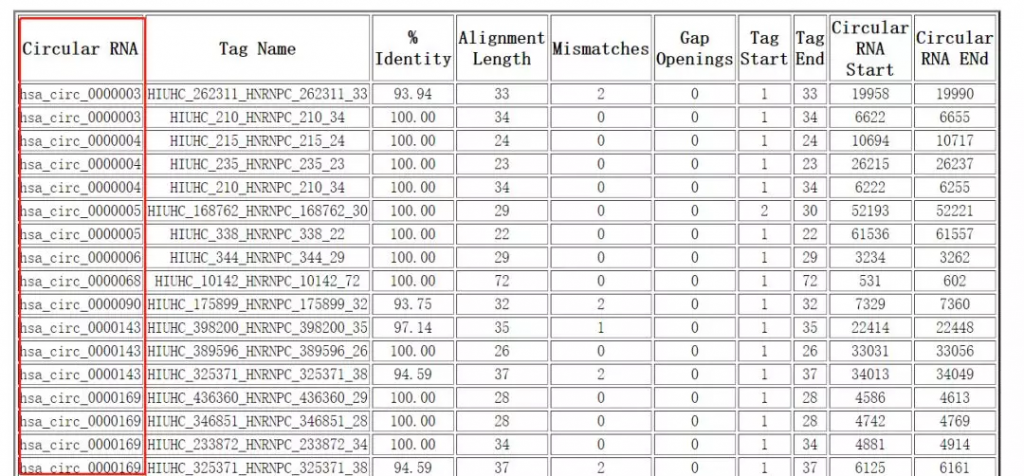
二、搜索circRNA结合的miRNA;
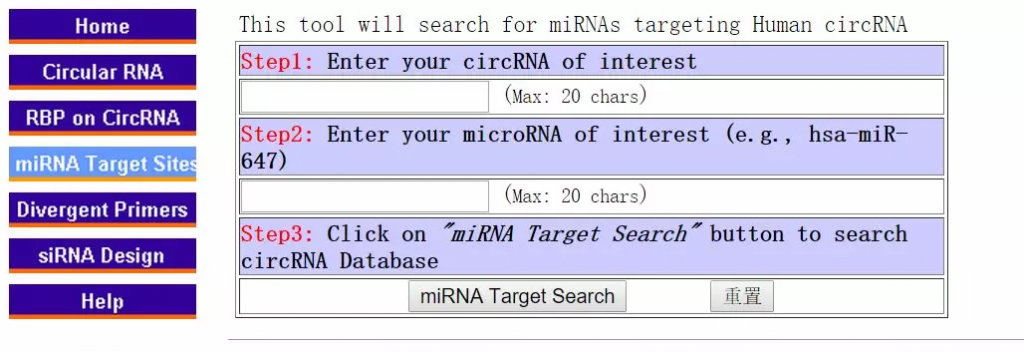
在右侧输入框中输入hsa_circ_0000234,然后单击miRNA target search,就可以看到预测到的miRNA了:
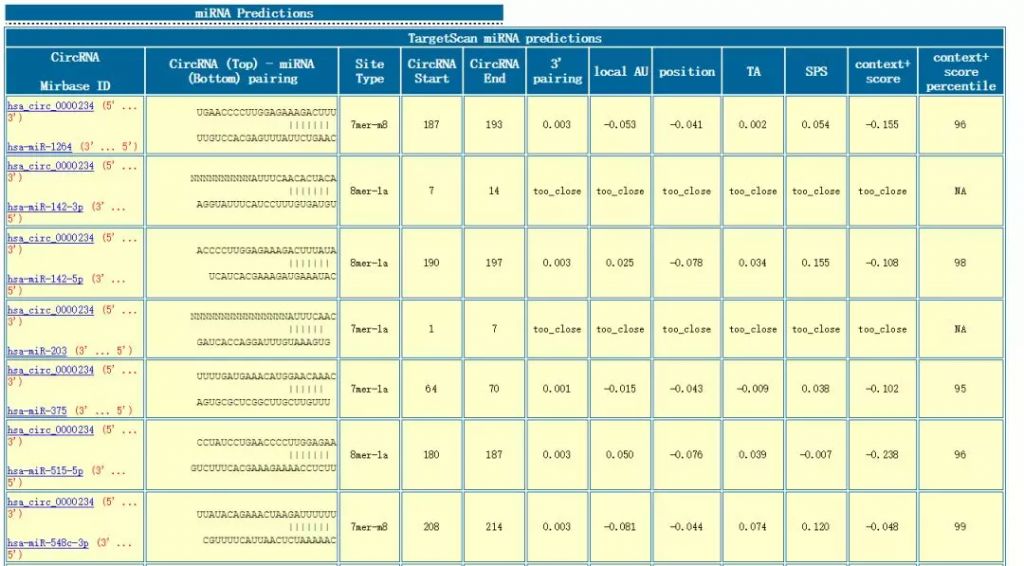
三、设计circRNA的引物及siRNA序列;
单击左侧的Divergent Primers:
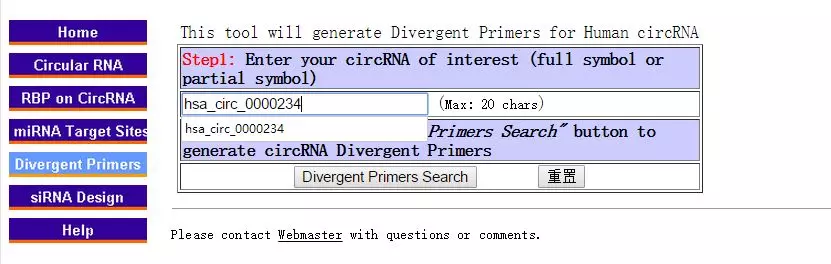
输入circRNA的名字后单击Divergent Primers Search:
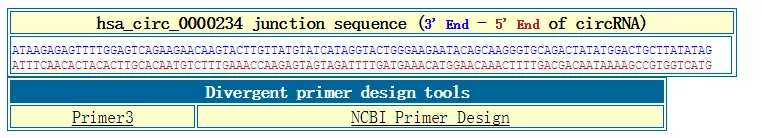
siRNA的设计:
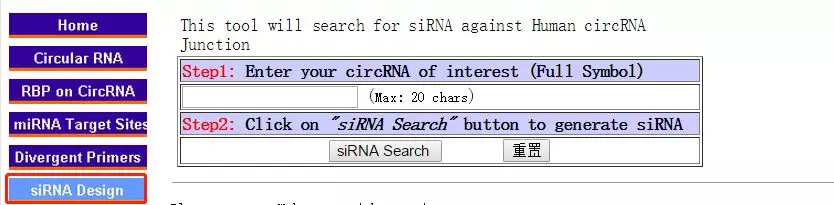
输入名字后单击siRNA search:

好,数据库就介绍到这里。
ref:Dudekula DB, Panda AC, Grammatikakis I, De S, Abdelmohsen K, and Gorospe M.CircInteractome: A web tool for exploring circular RNAs and their interacting proteins and microRNAs. RNA Biology, 2016, Jan 2;13(1):34-42





RegRNA2.0好像用不了了,是么/?
可以用啊